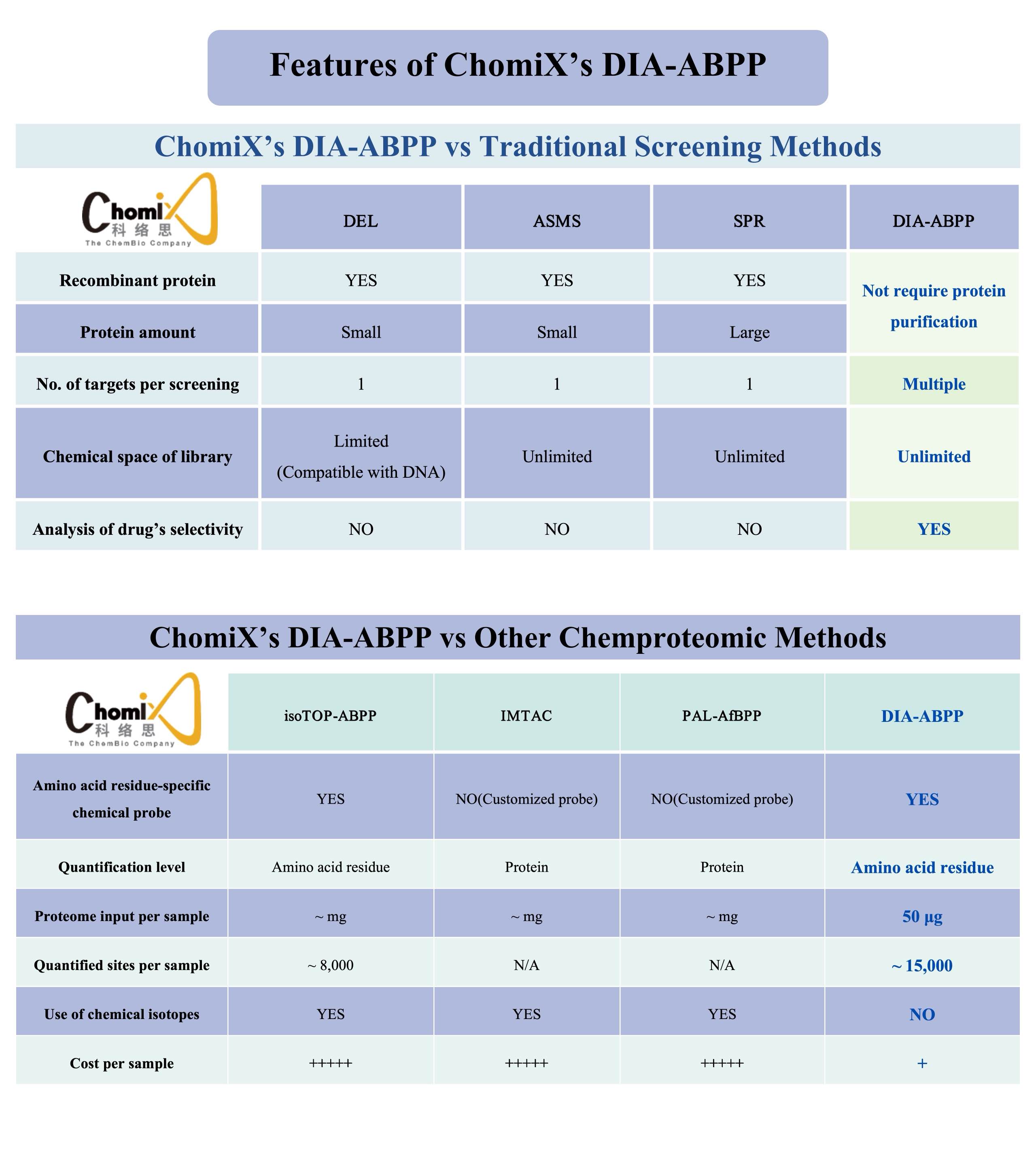
Unsere wissenschaftliche Plattform,ChomiX-Bildschirm, das auf DIA-ABPP (Data Independent Acquisition-Activity Based Protein Profiling) basiert, ist ein wichtiger Teil unserer ABPP-führenden Methodik. Durch unsereChomiX-BildschirmMithilfe unserer Technologieplattform können wir kovalente Leitverbindungen auf nicht behandelbare Ziele untersuchen. Der spezifische Prozess umfasst die Behandlung lebender Zellen mit kovalenten Molekülen, die Trennung und Anreicherung von mit chemischen Sonden markierten Peptiden, die Detektion mittels hochauflösender Massenspektrometrie und den Aufbau eines Netzwerks zur Bindung von Wirkstoff und Zielstelle. Dies ermöglicht es uns, die Bindungsfähigkeit und Selektivität von Arzneimitteln für bestimmte Aminosäureresttaschen auf mehreren Proteinzielen gleichzeitig zu bewerten und so Kandidatenverbindungen zu generieren. Derzeit haben wir eine umfassende Proteinzielbibliothek aufgebaut, einschließlich der größten inländischen Bibliothek kovalenter Verbindungen, die auf Cystein abzielen , basierend auf unserer chemischen Sondenbibliothek und anderen charakteristischen Kernmodulen.
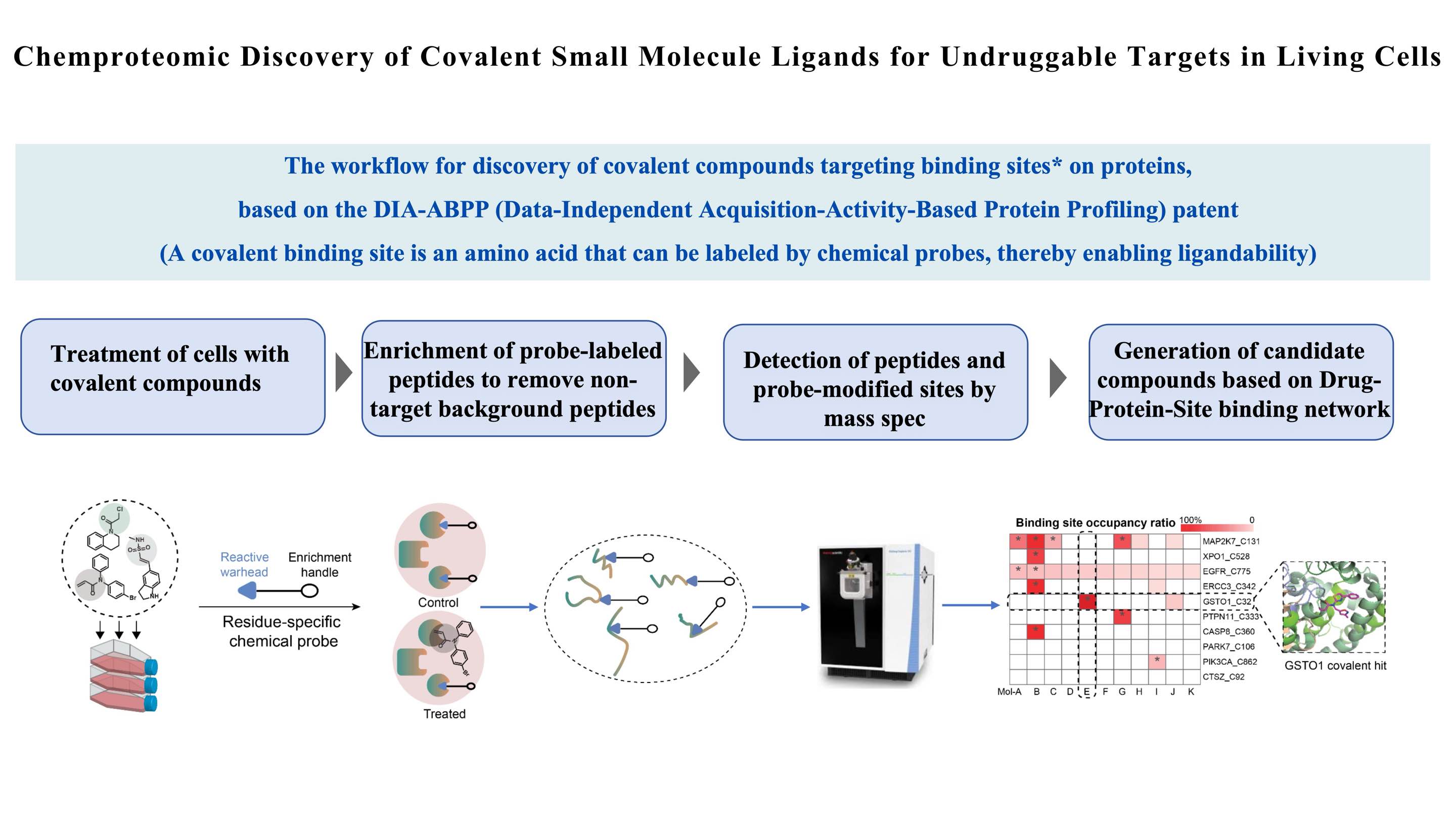
Die Belegungsrate der Bindungsstelle (Protein_Cys-Stelle) für jede Verbindung wird anhand der Massenspektrometerintensitäten quantifizierter sondenmarkierter Peptide bestimmt. Im Wesentlichen konkurriert die Cystein-spezifische Sonde mit Verbindungen um die Bindung an eine bestimmte Bindungstasche. Je niedriger das Signal des sondenmarkierten Peptids ist, desto stärker ist die Besetzungsfähigkeit der Verbindung. Für jede Cysteinstelle des Proteins von Interesse (POI) wird die Bestimmung der dosisabhängigen Zielbindungsverhältnisse (TE50) für ausgewählte Verbindungen durchgeführt. Hit-Kandidaten werden dann durch die Rangfolge ihrer TE50-Werte bestätigt. Je niedriger der TE50-Wert, desto stärker besetzt die Verbindung die Cysteinstelle des POI in lebenden Zellen.