
Produkte
Chemoproteomische Entdeckung neuer Leitstrukturen für nicht behandelbare Ziele
Technischer Hintergrund
Derzeit werden nur etwa 800 Proteine von von der FDA zugelassenen Medikamenten angegriffen, und eine große Anzahl krankheitsbezogener Ziele ist „nicht behandelbar“. Denn derzeit basieren die meisten Technologien auf gereinigten Proteinen. Das Aufkommen der Chemproteomik hat die Arzneimittelforschung von gereinigten Proteinen bis hin zu lebenden Zellen revolutioniert. Es ist in der Lage, die Wechselwirkungen zwischen kleinen Molekülen und Proteinen im menschlichen Proteommaßstab quantitativ zu analysieren. Nun wurde die Entdeckung kovalenter Strukturen gegen spezifische Aminosäurereste von Proteinzielen wie Cystein, Lysin, Methionin und Tyrosin in Zelllysaten und lebenden Zellen nachgewiesen. ChomiX wird seine eigene Chemoproteomik-Plattform nutzen, um die Barrieren „nicht behandelbarer“ Ziele schnell zu durchbrechen.

Technische Vorteile
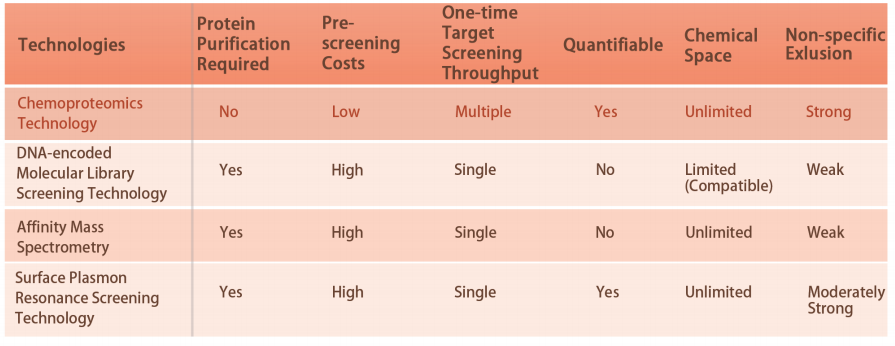
Technische Merkmale der Plattform
Die DIA-ABPP-Plattform dient der Entdeckung kovalenter Bindemittel, die Proteinziele durch kovalente Reaktionen mit der Thiolgruppe von Cysteinresten angreifen. Sobald aktive Cysteinreste von Proteinen bevorzugt durch kovalente Moleküle besetzt sind, wird die Markierung der universellen Sonde, die spezifisch mit der Thiolgruppe reagiert, konkurriert. In Kombination mit DIA-basierten quantitativen Proteomics-Techniken konnten die Intensitäten sondenmarkierter Peptide zwischen kovalentem Molekül und Kontrollgruppe quantifiziert werden. Diese Signalunterschiede stellen die Besetzungsraten kovalenter Moleküle gegenüber ihren Bindungsproteinen dar. Im Gegensatz zu herkömmlichen Single-Target-Screening-Strategien ermöglicht die DIA-ABPP-Plattform eine quantitative Analyse der Wechselwirkungen zwischen Fragment-Elektrophilen und nahezu allen Proteinen in lebenden Zellen mit der Auflösung von Aminosäureresten, was die Effizienz erheblich verbessert und das Risiko für das Projekt zur Entdeckung von Leitstrukturen mit hohem Durchsatz verringert .

Arbeitsablauf beim Screening kovalenter Leitverbindungen
Merkmal 1

Unsere arzneimittelähnliche Molekülbibliothek enthält eine Vielzahl reaktiver Gruppen (Gefechtsköpfe), die sich durch eine milde elektrophile Reaktivität auszeichnen, wie z. B. Acrylamid, Chloracetamid und Alkinamid. Diese Moleküle verfügen über strukturell unterschiedliche Pharmakophore, die der Fünferregel von Lipinski entsprechen.
Merkmal 2

Derzeit umfasst die Bibliothek der von der Thiol-spezifischen chemischen Sonde erfassten Proteinziele 39.962 Cysteinstellen von 12.421 Proteinen, einschließlich Kinase, Phosphatase, Ligasen und Transkriptionsfaktoren. Die Bibliothek wird ständig aktualisiert und gewährleistet so den Zugriff auf die neuesten Informationen zu bestimmten Zielen und Standorten. Detaillierte Informationen zu bestimmten Zielen und Standorten finden Sie über den unten stehenden Link.
Merkmal 3

In einem einzigen Screening-Experiment ist die Plattform in der Lage, den Angriff kleiner Moleküle (A2-H6) auf alle markierten Proteine im gesamten Proteom zu quantifizieren. Die obige Abbildung zeigt die Standortbelegung ausgewählter klinisch wichtiger Ziele durch kovalente Moleküle. Je dunkler die Farbe, desto höher ist die Besetzung der Bindungstaschen mit Molekülen, einschließlich orthosterischer und allosterischer Stellen (das Sternchen zeigt an, dass die Besetzungsrate höher als 80 % ist). Somit können die Affinität und Selektivität jedes Moleküls gleichzeitig zur anschließenden Strukturoptimierung bewertet werden.




