Nukleinsäure-Protein-Wechselwirkungen durch Proximity-Labeling-Strategie aufgedeckt: Erforschung von G4PID-Sonden und PLGPB-Strategie
In diesem Artikel wird eine innovative Studie vorgestellt, in der die Autoren eine neuartige bifunktionale Sonde namens G4PID vorstellen. Diese Sonde kombiniert die G4-Bindungsdomäne (RHAU23) von RHAU mit der miniTurbo-Biotinligase und ermöglicht so ein präzises Targeting von G-Quadruplexen (G4) und die Markierung von G4-interagierenden Proteinen in lebenden Zellen. G4-Strukturen, die aus Guanin-reichen Nukleinsäuresequenzen bestehen, sind von entscheidender Bedeutung für verschiedene zelluläre Prozesse, einschließlich Genregulation, DNA-Replikation und Reparatur. Obwohl die Bedeutung von G4 weithin anerkannt ist, sind die spezifischen Mechanismen innerhalb von Zellen und ihre Wechselwirkungen mit Proteinen noch weitgehend unerforscht. Mithilfe von G4PID entwickelten die Autoren eine spezielle Biotin-Markierungsmethode (PLGPB), um mit G4 interagierende Proteine genau zu identifizieren und zu analysieren und so Licht auf die entscheidende Rolle von G4-Strukturen bei Zellfunktionen zu werfen. Dieser Ansatz identifizierte erfolgreich 149 Proteinkandidaten, die mit G4 interagieren und hauptsächlich an der Transkriptionsregulation, dem mRNA-Spleißen und der Chromatin-Remodellierung beteiligt sind. Die Validierung von sieben Kandidatenproteinen ergab, dass sie die RNA-G4-Bindung bevorzugen, wenn auch mit unterschiedlichen Affinitäten gegenüber DNA-G4. Eine detaillierte Untersuchung des Spleißfaktors SF3B4 zeigte, dass er mit G4-Strukturen interagiert und alternative Spleißereignisse beeinflusst. Die Stabilisierung von G4-Strukturen veränderte die Bindungseffizienz von SF3B4 und beeinflusste das alternative Spleißen spezifischer Gene (z. B. INPPL1 und PPP6R2).

Berberin, ein traditionelles Alkaloid mit weitreichenden pharmakologischen Wirkungen, darunter Entzündungshemmung, Hypoglykämie und Herz-Kreislauf-Schutz, hat große Aufmerksamkeit erregt. Die genauen molekularen Mechanismen, insbesondere bei der Unterdrückung von Entzündungen, sind jedoch weiterhin unklar.
Diese Studie schließt diese Wissenslücke mithilfe der ABPP-Technologie, um EIF2AK2 als kritisches Ziel zu identifizieren, an dem Berberin aufgrund seiner entzündungshemmenden Wirkung beteiligt ist. Die Ergebnisse vertiefen unser Verständnis des Mechanismus von Berberin und liefern eine wissenschaftliche Grundlage für die Neupositionierung von Berberin und die Entwicklung neuer, auf EIF2AK2 ausgerichteter entzündungshemmender Medikamente.
Das Team nutzte fortschrittliche chemoproteomische Methoden, um die Wechselwirkungen von Berberin mit intrazellulären Proteinen systematisch zu untersuchen und seine spezifische Bindung an EIF2AK2 und die Modulation seiner enzymatischen Aktivität zu bestätigen. Dies beeinflusst die Entzündungsreaktionswege und hemmt wirksam das Fortschreiten der Entzündung. Dieser bedeutende Durchbruch bietet Einblicke in den entzündungshemmenden Mechanismus von Berberin und unterstützt die Entwicklung neuartiger Therapien gegen EIF2AK2.
ChomiX bietet hochmoderne Dienste wie ABPP und CETSA, um Forscher bei der Erforschung von Arzneimittelmechanismen und der Beschleunigung neuer Arzneimittelentwicklungsbemühungen zu unterstützen.
Forschungsroute

Experimenteller Prozess
1. Konstruktion und Spezifitätsüberprüfung der G4PID-Sonde.
Die Autoren konstruierten zunächst die G4PID-Sonde mit der G4-Bindungsdomäne RHAU23 am N-Terminus und dem miniTurbo am C-Terminus, verbunden durch einen flexiblen Linker. Expression und Reinigung erfolgten mit dem Stamm BL21 (DE3). Spezifische Bindungsvalidierungsexperimente mit gereinigtem G4PID zeigten dessen präzise Bindung an BCL2 G4 mit einem Kd-Wert von 15 ± 7 nM und zeigten gleichzeitig eine vernachlässigbare Affinität zu anderen Nicht-G4-Strukturen. Darüber hinaus bewerteten die Autoren die Biotinylierungsspezifität von G4PID und zeigten seine Fähigkeit, G4-interagierende Proteine selektiv zu biotinylieren.
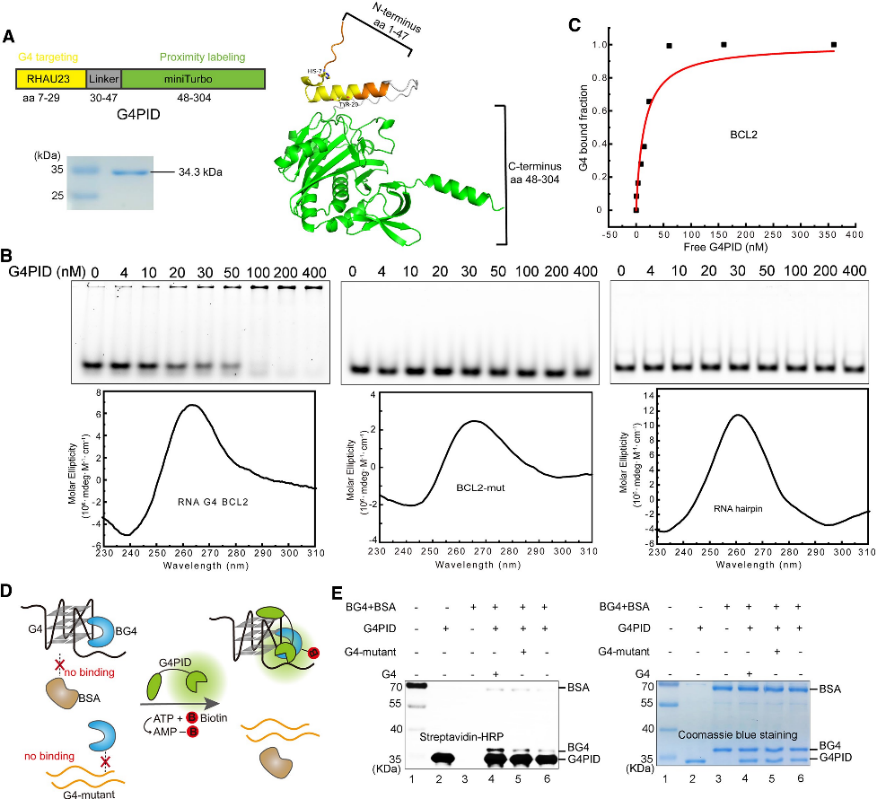
Abbildung 1 G4 PID vermittelt die spezifische Biotinylierung des G 4-interagierenden Proteins
2. RNA-G4-Bindungsfähigkeit von G4PID in Zellen und Biotin-Markierungseigenschaften.
Die Autoren etablierten HA-G4PID- und HA-miniTurbo-HeLa-Zelllinien, um die Bindungsaffinität von G4PID an intrazelluläre RNA G4 über eCLIP-Experimente zu untersuchen. Die Ergebnisse belegen die selektive Wechselwirkung von G4PID mit RNA G4 und zeigen im Vergleich zu Zufallssequenzen eine deutlich höhere Bindungsaffinität zu RNA-Sequenzen, die PQS enthalten. Darüber hinaus beobachteten die Autoren einzigartige Merkmale von G4PID, die die Einleitung der Biotinmarkierung innerhalb von Zellen erleichtern, ohne dass Cofaktoren erforderlich sind. Dies führte zu einer erheblichen Verbesserung der Proteinmarkierungseffizienz, wobei innerhalb von 30 Minuten deutliche Biotinylierungssignale für G4PID erkannt wurden, was auf eine schnellere Markierungskinetik im Vergleich zu miniTurbo hinweist.
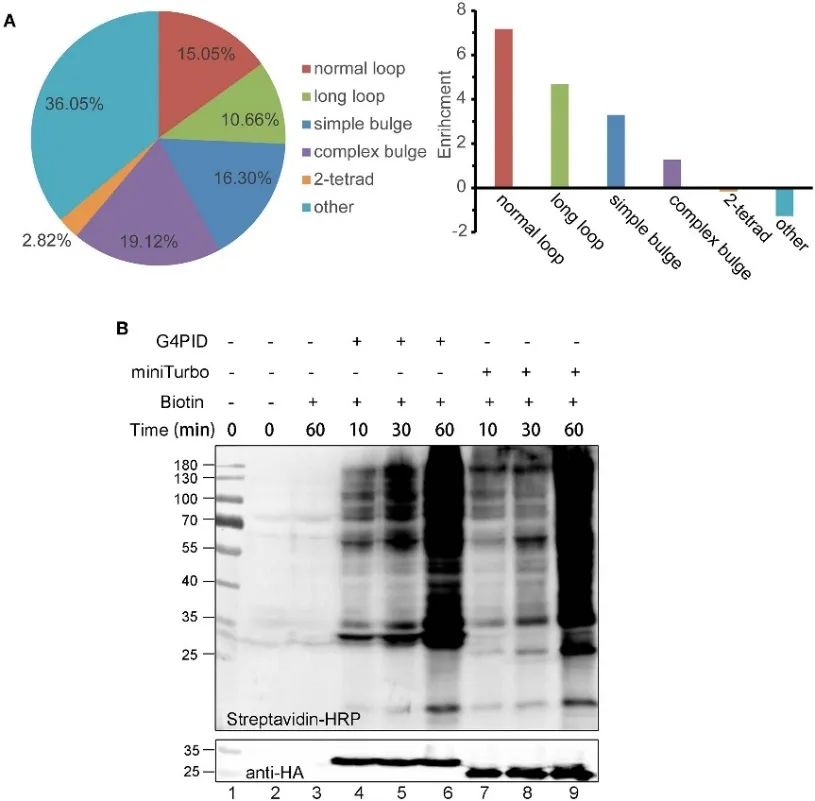
Abbildung 2 G 4 PID kombiniert mit RNA G4-Experimenten und Optimierung für die Markierungseffizienz in HEK293T-Zellen.
3. Erfassung und Erkennung des intrazellulären G4-interagierenden Proteoms mithilfe der PLGPB-Methode.
Anschließend verwendeten die Autoren die G4PID-Sonde, um das mit G4 interagierende Proteom in lebenden Zellen mithilfe des Proximity Labeling of G4-Interacting Proteins (PLGPB)-Ansatzes zu erfassen und nachzuweisen. In drei unabhängigen Wiederholungsexperimenten mit 578 Proteinen, bei denen eine Identität von 80 % erreicht wurde, wurde eine Transfektion mit G4PID und miniTurbo in HEK293T-Zellen durchgeführt. Das Screening ergab 149 Kandidatenproteine, die mit G4 interagieren, und zeigte eine signifikante Überschneidung mit bestehenden Datenbanken von G4-interagierenden Proteinen. Darüber hinaus deutete die Anreicherungsanalyse der Gene Ontology (GO) auf einen erheblichen Zusammenhang dieser Proteine mit der Transkriptionsregulation, dem mRNA-Spleißen und der Chromatin-Remodellierung hin, was mit früheren Erkenntnissen der G4-Forschung übereinstimmt.
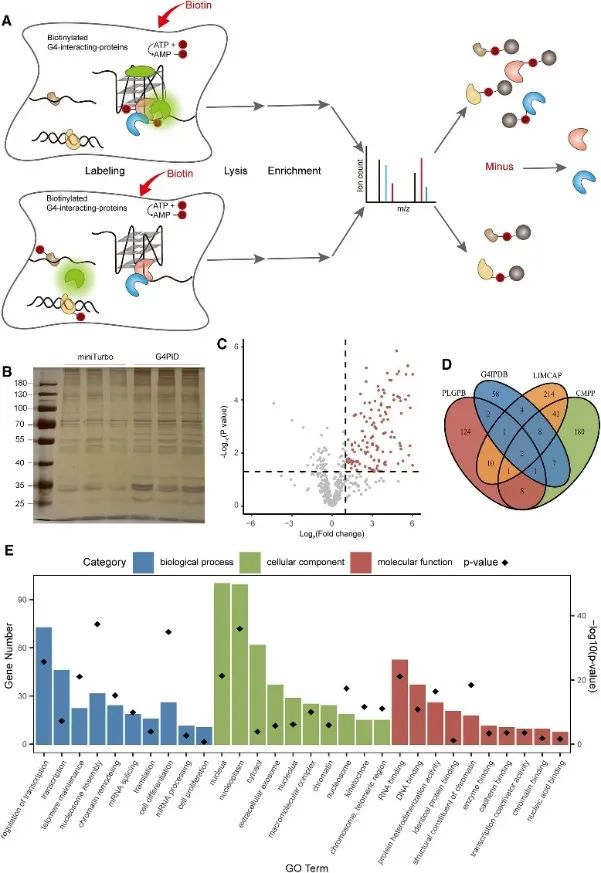
Abbildung 3 identifizierte die Proteine, die mit G4 mit der PLGPB-Methode interagieren.
4. Validierung der G4-Bindungsaffinität bei neu identifizierten Kandidaten.
Mithilfe der PLGPB-Methode entdeckten die Autoren eine Reihe von Kandidaten, die verschiedene Funktionskategorien umfassen, darunter häufige Telomer-assoziierte Proteine, Transkriptionsfaktoren und relativ seltene proteinbindende Proteine. Dies weist darauf hin, dass die PLGPB-Methode nicht nur zuvor anerkannte G4-interagierende Proteine bestätigt, sondern auch die Proteinlandschaft erheblich erweitert, indem sie neue Kandidaten entdeckt, die verschiedene Funktionsklassen umfassen. Darüber hinaus validierten die Autoren die Bindungskapazität dieser Kandidaten an RNA G4 und DNA G4 und zeigten eine ausgeprägte Präferenz für RNA G4 und Variationen in der Affinität gegenüber DNA G4 in verschiedenen Konformationen. Diese Ergebnisse unterstreichen das Potenzial, die Protein-G4-Interaktion durch Regulierung der G4-Konformation zu modulieren.
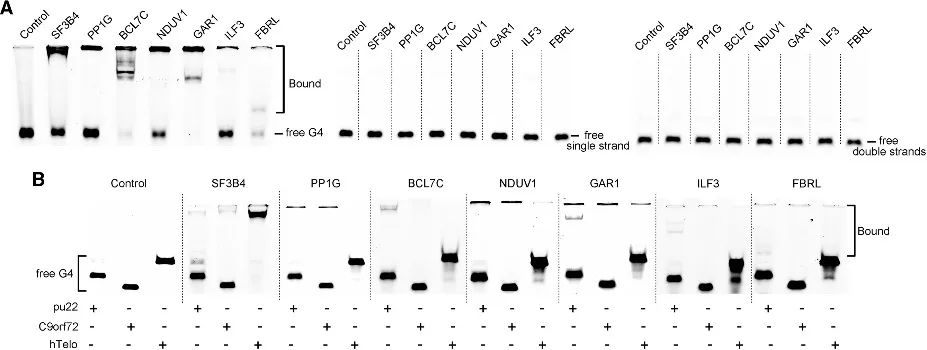
Abbildung 4 Interaktion zwischen den Kandidatenproteinen und RNA G4 und DNA G4.
5. Bestimmung hochkonfidenter Bindungsstellen der SF3B4- und G4-Struktur.
Während der Untersuchung des In-vivo-Modus der G4-Bindung entdeckten die Autoren die bedeutende Rolle des SF3B4-Proteins. Die Bindungsstellen von SF3B4, das für seine Beteiligung am RNA-Spleißen bekannt ist, wurden durch eCLIP-Experimente und eine umfassende Analyse der rG4-seq- und DMS-seq-Daten identifiziert. Ungefähr die Hälfte dieser Bindungsstellen enthielt PQS, überwiegend assoziiert mit unkonventionellen G4-Strukturen. Darüber hinaus beobachteten die Autoren eine enge räumliche Beziehung zwischen SF3B4-Bindungsstellen und RNA-PQS, mit einer angereicherten Verteilung hauptsächlich innerhalb intronischer Regionen, insbesondere in der Nähe der 3'-Spleißstelle. Diese Ergebnisse deuten stark darauf hin, dass die Wechselwirkung zwischen SF3B4 und der G4-Struktur eine entscheidende Rolle bei alternativen Spleißmechanismen spielen könnte.
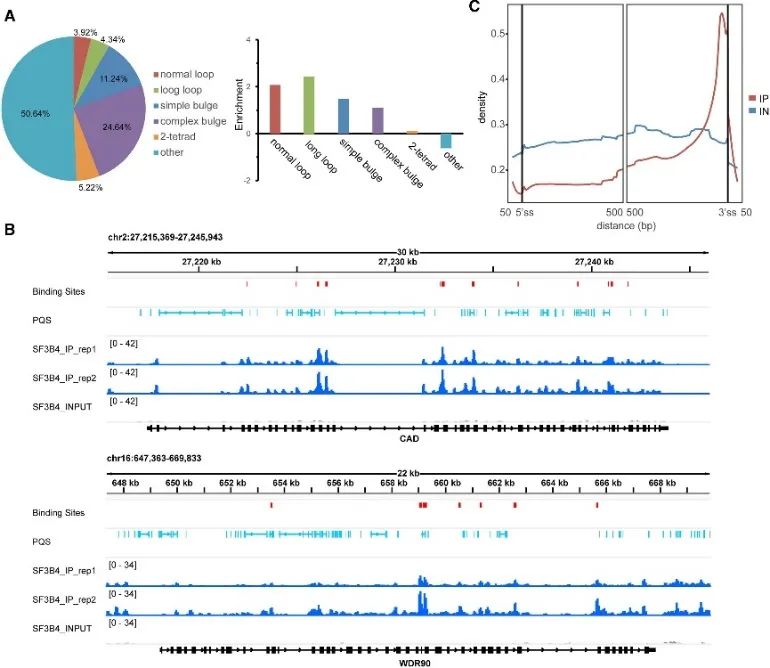
Abbildung 5 identifiziert die G4-Bindungspräferenz von SF3B4 in Zellen mithilfe der eCLIP-Technologie.
6. Die G4-Struktur reguliert das SF3B4-vermittelte alternative Spleißen.
Die Autoren führten eine Analyse der SF3B4-Bindungsstellen durch, um dessen Zusammenhang mit der G4-Struktur aufzudecken, und untersuchten weiter seinen Regulierungsmechanismus bei alternativen Spleißereignissen. Herkömmliche PQS, die am alternativen Spleißen beteiligt sind, wurden gescreent, wobei zwei Gene, INPPL1 und PPP6R2, für eine detaillierte Untersuchung ausgewählt wurden. G4-Strukturen innerhalb von INPPL1 und PPP6R2, die als G4-INPPL1 und G4-PPP6R2 bezeichnet werden, wurden bestätigt, und es wurde festgestellt, dass SF3B4 spezifisch an diese G4-Strukturen bindet. Darüber hinaus wurde gezeigt, dass die Zugabe des G4-Liganden Pyridostatin (PDS) die SF3B4-Bindung an G4-Strukturen verringert und folglich Exon-Einschlussereignisse während des INPPL1- und PPP6R2-mRNA-Spleißens fördert. Diese Ergebnisse weisen auf die potenzielle Bedeutung der G4-Struktur beim SF3B4-vermittelten alternativen Spleißen hin.
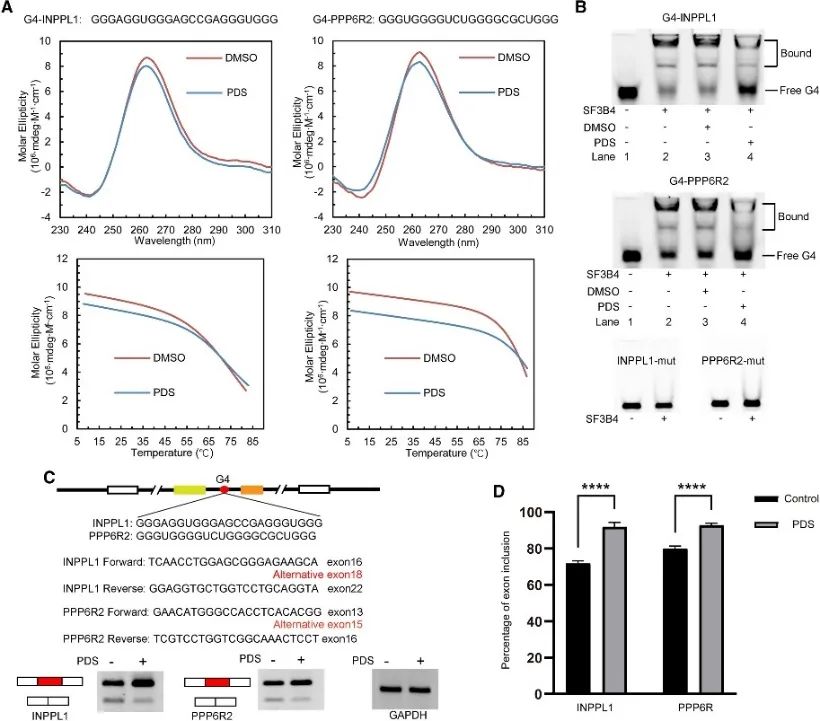
Abbildung 6 SF3B4 bindet die G4-Struktur und beeinflusst das alternative Spleißen der mRNA.
Zusammenfassend stellt diese Studie ein effizientes Toolkit vor, das die G4PID-Sonde und die PLGPB-Strategie umfasst und Aufschluss darüber gibt, wie sich das Zusammenspiel zwischen G4 und Proteinen innerhalb der Zelle auf die Genexpression auswirkt und insbesondere den entscheidenden Prozess des alternativen Spleißens beeinflusst. Diese Erkenntnisse vertiefen unser Verständnis der regulatorischen Mechanismen, die G4 an zellulären biologischen Prozessen beteiligt, und ebnen den Weg für die weitere Erforschung therapeutischer Strategien für G4-bedingte Krankheiten.

