Interacciones ácido nucleico-proteína reveladas mediante la estrategia de etiquetado de proximidad: exploración de las sondas G4PID y la estrategia PLGPB
Este artículo presenta un estudio innovador en el que los autores presentan una nueva sonda bifuncional denominada G4PID. Esta sonda combina el dominio de unión a G4 (RHAU23) de RHAU con la biotina ligasa miniTurbo, lo que permite apuntar con precisión a los cuadruplex G (G4) y etiquetar las proteínas que interactúan con G4 dentro de las células vivas. Las estructuras G4, formadas por secuencias de ácidos nucleicos ricas en guanina, son fundamentales en diversos procesos celulares, incluida la regulación genética, la replicación y la reparación del ADN. A pesar del reconocimiento generalizado de la importancia del G4, los mecanismos específicos dentro de las células y sus interacciones con las proteínas siguen en gran medida inexplorados. Empleando G4PID, los autores desarrollaron un método de etiquetado de biotina especializado (PLGPB) para identificar y analizar con precisión las proteínas que interactúan con G4, arrojando luz sobre el papel crucial de las estructuras de G4 en las funciones celulares. Este enfoque identificó con éxito 149 proteínas candidatas que interactúan con G4, predominantemente involucradas en la regulación transcripcional, el empalme de ARNm y la remodelación de la cromatina. La validación de siete proteínas candidatas reveló su preferencia por la unión del ARN G4, aunque con diferentes afinidades hacia el ADN G4. El examen detallado del factor de empalme SF3B4 demostró su interacción con las estructuras G4, lo que afecta los eventos de empalme alternativos. La estabilización de las estructuras G4 alteró la eficiencia de unión de SF3B4 e influyó en el corte y empalme alternativo de genes específicos (p. ej., INPPL1 y PPP6R2).

La berberina, un alcaloide tradicional con una amplia gama de efectos farmacológicos que incluyen antiinflamatorios, hipoglucemia y protección cardiovascular, ha atraído considerable atención. Sin embargo, sus mecanismos moleculares precisos, particularmente en la supresión de la inflamación, aún no están claros.
Este estudio llena este vacío de conocimiento utilizando la tecnología ABPP para identificar EIF2AK2 como un objetivo crítico de la berberina por su acción antiinflamatoria. Los hallazgos profundizan nuestra comprensión del mecanismo de la berberina y proporcionan una base científica para reposicionar la berberina y desarrollar nuevos fármacos antiinflamatorios dirigidos a EIF2AK2.
El equipo empleó métodos quimioproteómicos avanzados para investigar sistemáticamente las interacciones de la berberina con proteínas intracelulares, confirmando su unión específica a EIF2AK2 y la modulación de su actividad enzimática. Esto influye en las vías de respuesta inflamatoria, inhibiendo eficazmente la progresión de la inflamación. Este importante avance ofrece información sobre el mecanismo antiinflamatorio de la berberina y respalda el desarrollo de nuevas terapias dirigidas a EIF2AK2.
ChomiX ofrece servicios de vanguardia como ABPP y CETSA para ayudar a los investigadores a explorar los mecanismos de los fármacos y acelerar los esfuerzos de desarrollo de nuevos fármacos.
Ruta de investigación

Proceso experimental
1. Construcción y Verificación de Especificidad de la Sonda G4PID.
Inicialmente, los autores diseñaron la sonda G4PID, con el dominio de unión a G4 RHAU23 colocado en el extremo N y el miniTurbo en el extremo C, unidos por un conector flexible. La expresión y purificación se llevaron a cabo utilizando la cepa BL21 (DE3). Los experimentos de validación de unión específica con G4PID purificado revelaron su unión precisa a BCL2 G4, mostrando un valor de Kd de 15 ± 7 nM, mientras que mostraba una afinidad insignificante hacia otras estructuras que no son G4. Además, los autores evaluaron la especificidad de biotinilación de G4PID, demostrando su capacidad para biotinilar selectivamente proteínas que interactúan con G4.
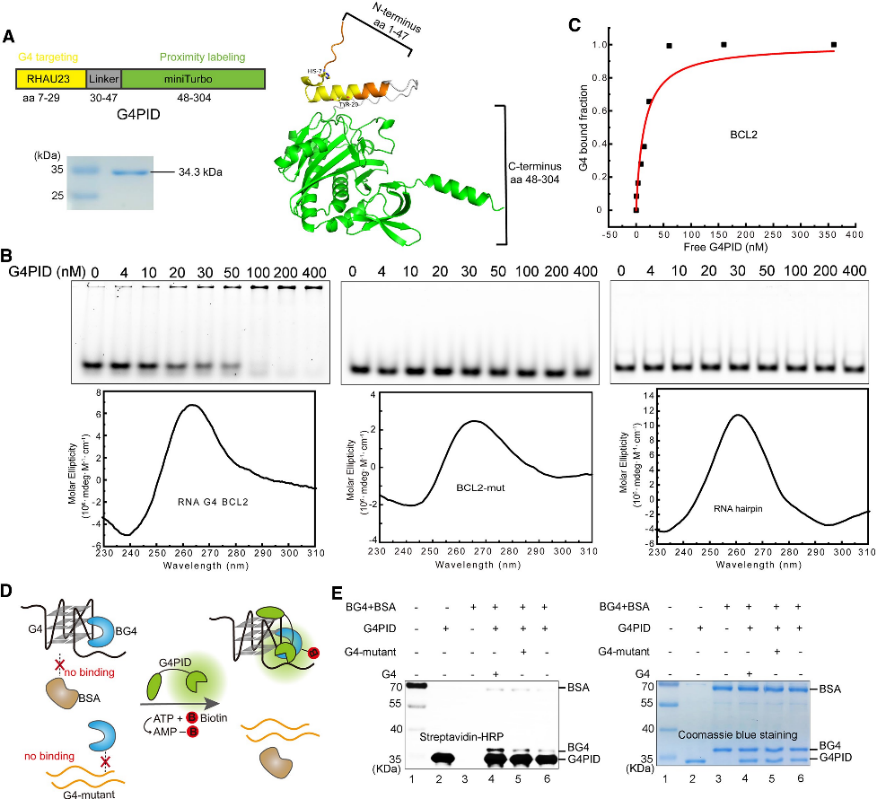
Figura 1 G4 PID media la biotinilación específica de la proteína que interactúa con G 4
2. Capacidad de unión al ARN G4 de G4PID en células y características de etiquetado con biotina.
Los autores establecieron líneas celulares HA-G4PID y HA-miniTurbo HeLa para investigar la afinidad de unión de G4PID al ARN G4 intracelular mediante experimentos eCLIP. Los resultados demuestran la interacción selectiva de G4PID con el ARN G4, mostrando una afinidad de unión notablemente mayor a las secuencias de ARN que contienen PQS en comparación con las secuencias aleatorias. Además, los autores observaron características únicas de G4PID que facilitan el inicio del etiquetado con biotina dentro de las células sin requerir cofactores. Esto condujo a una mejora significativa en la eficiencia del etiquetado de proteínas, con señales de biotinilación evidentes detectadas para G4PID en 30 minutos, lo que indica una cinética de etiquetado más rápida en comparación con miniTurbo.
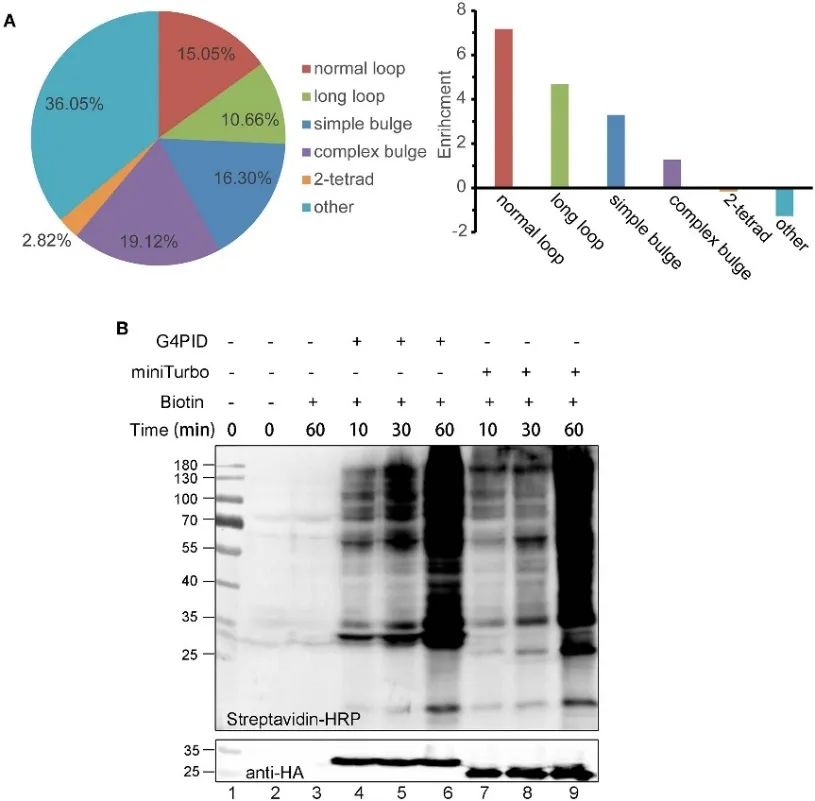
Figura 2 G 4 PID combinado con experimentos de ARN G4 y optimización para la eficiencia del etiquetado en células HEK293T.
3. Captura y detección del proteoma intracelular que interactúa con el G4 mediante el método PLGPB.
Posteriormente, los autores emplearon la sonda G4PID para capturar y detectar el proteoma que interactúa con G4 dentro de células vivas utilizando el método de etiquetado de proximidad de proteínas que interactúan con G4 (PLGPB). En tres experimentos replicados independientes que abarcaron 578 proteínas, logrando una identidad del 80%, se realizó la transfección con G4PID y miniTurbo en células HEK293T. El análisis reveló 149 proteínas candidatas que interactúan con G4, lo que muestra una superposición significativa con las bases de datos existentes de proteínas que interactúan con G4. Además, el análisis de enriquecimiento de Gene Ontology GO indicó una asociación sustancial de estas proteínas con la regulación transcripcional, el empalme de ARNm y la remodelación de la cromatina, en consonancia con hallazgos previos en la investigación de G4.
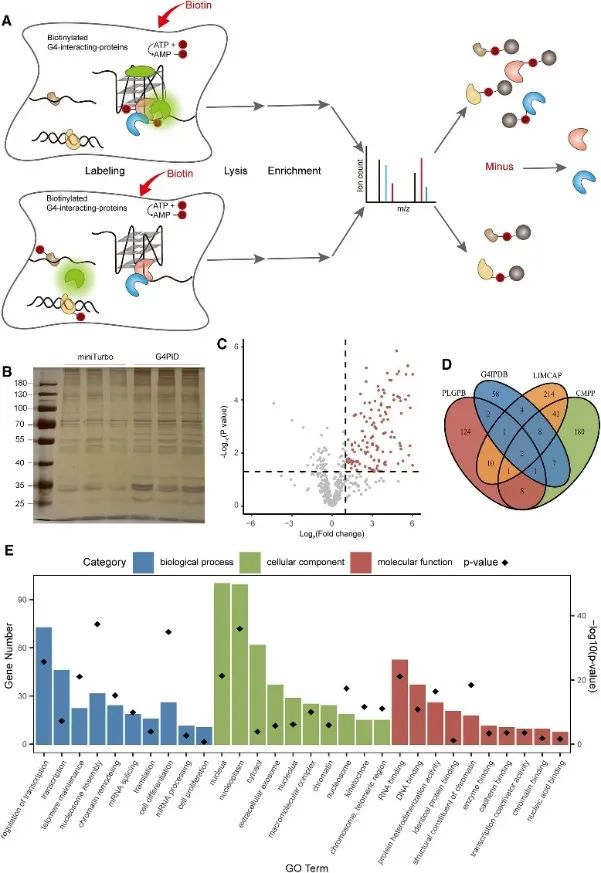
La Figura 3 identificó las proteínas que interactúan con G4 mediante el método PLGPB.
4. Validación de la afinidad vinculante de G4 en candidatos recién identificados.
A través del método PLGPB, los autores descubrieron una variedad de candidatos que abarcan varias categorías funcionales, incluidas proteínas comunes asociadas a los telómeros, factores de transcripción y proteínas de unión a proteínas relativamente poco comunes. Esto indica que el método PLGPB no solo reafirma las proteínas que interactúan con G4 previamente reconocidas, sino que también amplía significativamente el panorama de proteínas al descubrir nuevos candidatos que abarcan diversas clases funcionales. Además, los autores validaron la capacidad de unión de estos candidatos al ARN G4 y al ADN G4, revelando una marcada preferencia por el ARN G4 y variaciones en la afinidad hacia el ADN G4 en diferentes conformaciones. Estos hallazgos subrayan el potencial de modular la interacción proteína-G4 mediante la regulación de la conformación de G4.
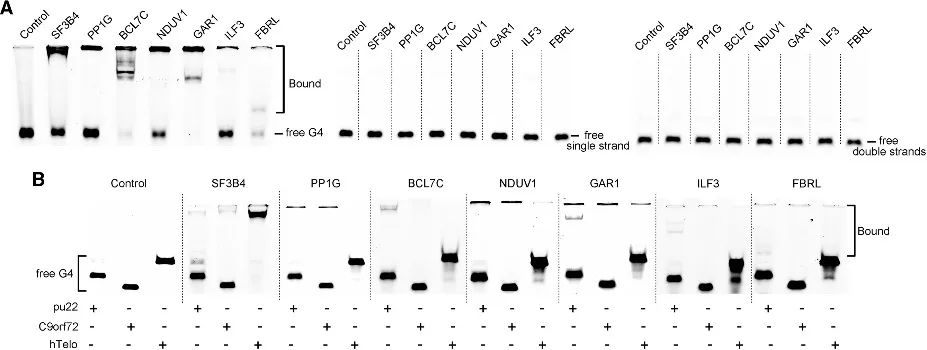
Figura 4 Interacción entre las proteínas candidatas y el ARN G4 y el ADN G4.
5. Determinación de los sitios de unión de alta confianza de la estructura SF3B4 y G4.
Durante la investigación sobre el modo in vivo de unión de G4, los autores descubrieron el importante papel de la proteína SF3B4. SF3B4, conocido por su participación en el empalme de ARN, identificó sus sitios de unión de alta confianza mediante experimentos eCLIP y un análisis exhaustivo de los datos de rG4-seq y DMS-seq. Aproximadamente la mitad de estos sitios de unión contenían PQS, predominantemente asociado con estructuras G4 no convencionales. Además, los autores observaron una estrecha relación espacial entre los sitios de unión de SF3B4 y el ARN PQS, con una distribución enriquecida principalmente dentro de regiones intrónicas, particularmente proximales al sitio de empalme 3'. Estos hallazgos sugieren fuertemente que la interacción entre SF3B4 y la estructura G4 podría desempeñar un papel crucial en mecanismos de empalme alternativos.
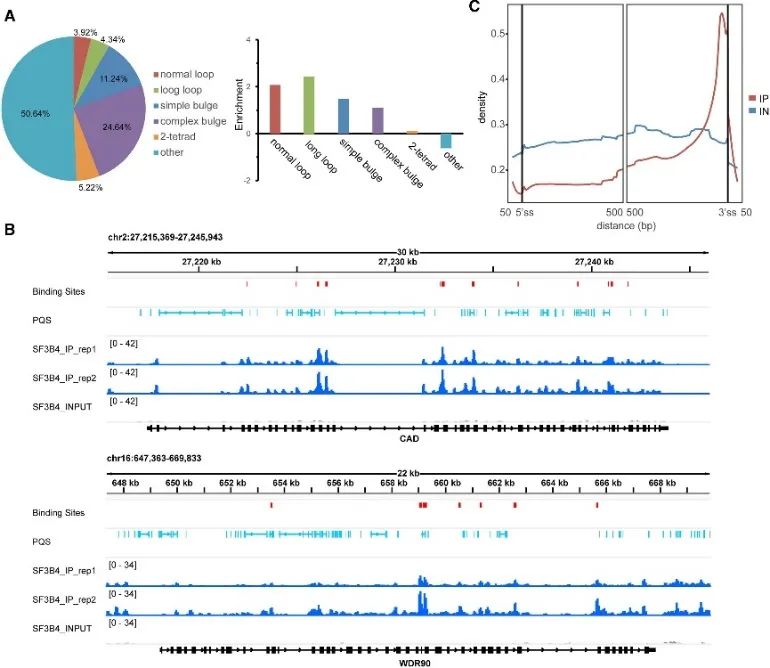
La Figura 5 identifica la preferencia de unión a G4 de SF3B4 en células que utilizan tecnología eCLIP.
6. La estructura G4 regula el empalme alternativo mediado por SF3B4.
Los autores realizaron un análisis de los sitios de unión de SF3B4 para revelar su asociación con la estructura G4 e investigaron más a fondo su mecanismo regulador en eventos de empalme alternativos. Se examinaron los PQS convencionales implicados en el empalme alternativo, y se seleccionaron dos genes, INPPL1 y PPP6R2, para un examen detallado. Se confirmaron las estructuras G4 dentro de INPPL1 y PPP6R2, denominadas G4-INPPL1 y G4-PPP6R2, y se identificó que SF3B4 se une específicamente a estas estructuras G4. Además, se demostró que la adición del ligando G4 piridostatina (PDS) disminuye la unión de SF3B4 a las estructuras G4, lo que promueve eventos de inclusión de exones durante el empalme del ARNm de INPPL1 y PPP6R2. Estos hallazgos indican la importancia potencial de la estructura G4 en el empalme alternativo mediado por SF3B4.
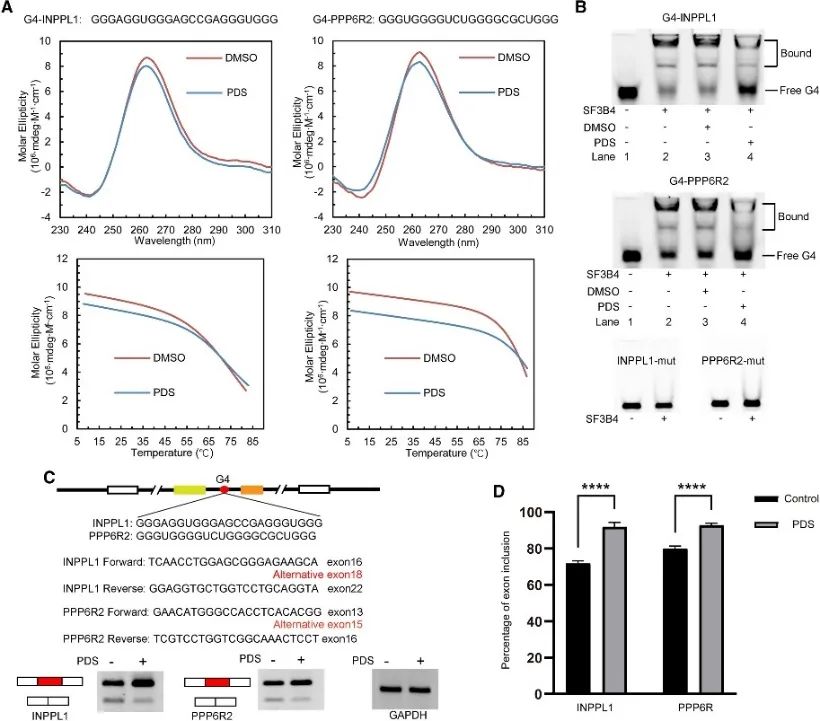
Figura 6 SF3B4 se une a la estructura G4 y afecta el empalme alternativo del ARNm.
En conclusión, este estudio presenta un conjunto de herramientas eficiente que comprende la sonda G4PID y la estrategia PLGPB, lo que arroja luz sobre cómo la interacción entre G4 y las proteínas dentro de la célula afecta la expresión génica, influyendo particularmente en el proceso fundamental de empalme alternativo. Estos hallazgos profundizan nuestra comprensión de los mecanismos reguladores que involucran al G4 en los procesos biológicos celulares y allanan el camino para una mayor exploración de estrategias terapéuticas para las enfermedades relacionadas con el G4.

