Interactions acide nucléique-protéine révélées grâce à la stratégie d'étiquetage de proximité : exploration des sondes G4PID et de la stratégie PLGPB
Cet article présente une étude innovante dans laquelle les auteurs introduisent une nouvelle sonde bifonctionnelle appelée G4PID. Cette sonde combine le domaine de liaison G4 (RHAU23) de RHAU avec la biotine ligase miniTurbo, permettant un ciblage précis des quadruplexes G (G4) et le marquage des protéines interagissant avec G4 dans les cellules vivantes. Les structures G4, formées de séquences d'acides nucléiques riches en guanine, jouent un rôle essentiel dans divers processus cellulaires, notamment la régulation des gènes, la réplication de l'ADN et la réparation. Malgré une large reconnaissance de l'importance du G4, les mécanismes spécifiques au sein des cellules et leurs interactions avec les protéines restent largement inexplorés. En utilisant G4PID, les auteurs ont développé une méthode spécialisée de marquage de la biotine (PLGPB) pour identifier et analyser avec précision les protéines interagissant avec G4, mettant ainsi en lumière le rôle crucial des structures G4 dans les fonctions cellulaires. Cette approche a permis d'identifier 149 protéines candidates interagissant avec G4, principalement impliquées dans la régulation transcriptionnelle, l'épissage de l'ARNm et le remodelage de la chromatine. La validation de sept protéines candidates a révélé leur préférence pour la liaison à l'ARN G4, bien qu'avec des affinités variables pour l'ADN G4. Un examen détaillé du facteur d'épissage SF3B4 a démontré son interaction avec les structures G4, impactant les événements d'épissage alternatifs. La stabilisation des structures G4 a modifié l'efficacité de liaison de SF3B4 et influencé l'épissage alternatif de gènes spécifiques (par exemple, INPPL1 et PPP6R2).

La berbérine, un alcaloïde traditionnel aux effets pharmacologiques étendus, notamment anti-inflammatoires, hypoglycémiques et protecteurs cardiovasculaires, a attiré une attention considérable. Cependant, ses mécanismes moléculaires précis, notamment dans la suppression de l’inflammation, restent flous.
Cette étude comble ce manque de connaissances en utilisant la technologie ABPP pour identifier EIF2AK2 comme cible critique engagée par la berbérine pour son action anti-inflammatoire. Les résultats approfondissent notre compréhension du mécanisme de la berbérine et fournissent une base scientifique pour repositionner la berbérine et développer de nouveaux médicaments anti-inflammatoires ciblés sur EIF2AK2.
L'équipe a utilisé des méthodes chimioprotéomiques avancées pour étudier systématiquement les interactions de la berbérine avec les protéines intracellulaires, confirmant ainsi sa liaison spécifique à EIF2AK2 et la modulation de son activité enzymatique. Cela influence les voies de réponse inflammatoire, inhibant efficacement la progression de l’inflammation. Cette avancée significative offre un aperçu du mécanisme anti-inflammatoire de la berbérine et soutient le développement de nouveaux traitements ciblant EIF2AK2.
ChomiX fournit des services de pointe comme ABPP et CETSA pour aider les chercheurs à explorer les mécanismes des médicaments et à accélérer les efforts de développement de nouveaux médicaments.
Itinéraire de recherche

Processus expérimental
1. Construction et vérification de spécificité de la sonde G4PID.
Les auteurs ont initialement conçu la sonde G4PID, avec le domaine de liaison G4 RHAU23 positionné à l'extrémité N-terminale et le miniTurbo à l'extrémité C, liés par un lieur flexible. L'expression et la purification ont été réalisées à l'aide de la souche BL21 (DE3). Des expériences de validation de liaison spécifiques avec le G4PID purifié ont révélé sa liaison précise à BCL2 G4, présentant une valeur Kd de 15 ± 7 nM, tout en affichant une affinité négligeable envers d'autres structures non G4. En outre, les auteurs ont évalué la spécificité de biotinylation du G4PID, démontrant sa capacité à biotinyler sélectivement les protéines interagissant avec le G4.
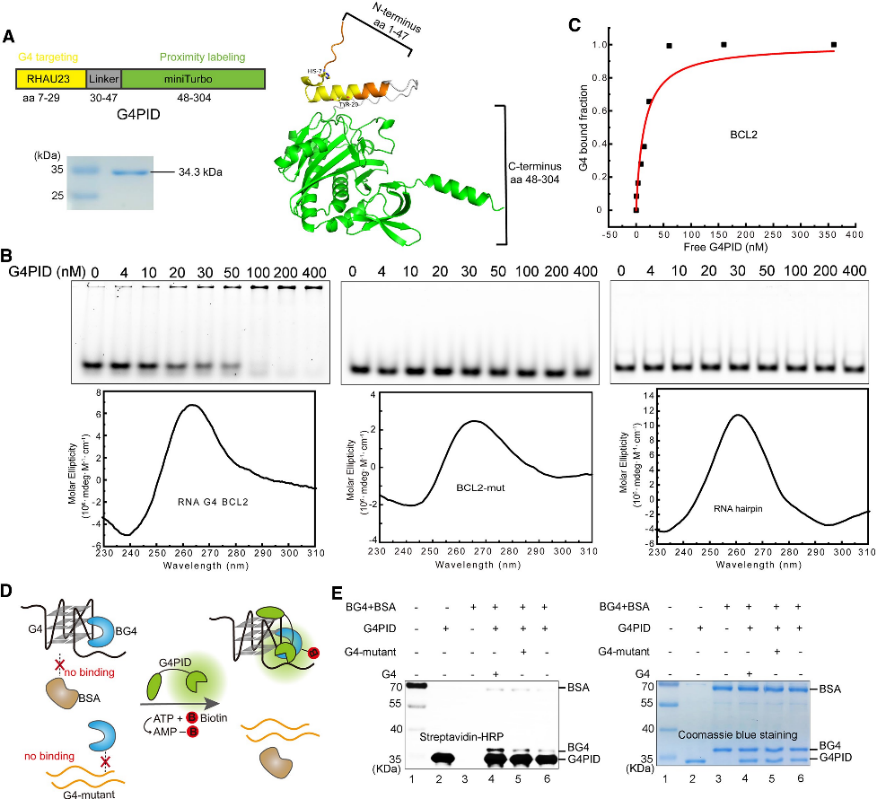
Figure 1 G4 PID médie la biotinylation spécifique de la protéine interagissant avec G 4
2. Capacité de liaison à l’ARN G4 du G4PID dans les cellules et caractéristiques de marquage de la biotine.
Les auteurs ont établi des lignées cellulaires HA-G4PID et HA-miniTurbo HeLa pour étudier l'affinité de liaison du G4PID à l'ARN intracellulaire G4 via des expériences eCLIP. Les résultats démontrent l'interaction sélective du G4PID avec l'ARN G4, présentant une affinité de liaison nettement plus élevée pour les séquences d'ARN contenant du PQS par rapport aux séquences aléatoires. De plus, les auteurs ont observé des caractéristiques uniques du G4PID facilitant l’initiation du marquage de la biotine dans les cellules sans nécessiter de cofacteurs. Cela a conduit à une amélioration significative de l’efficacité du marquage des protéines, avec des signaux de biotinylation évidents détectés pour le G4PID dans les 30 minutes, indiquant une cinétique de marquage plus rapide que celle du miniTurbo.
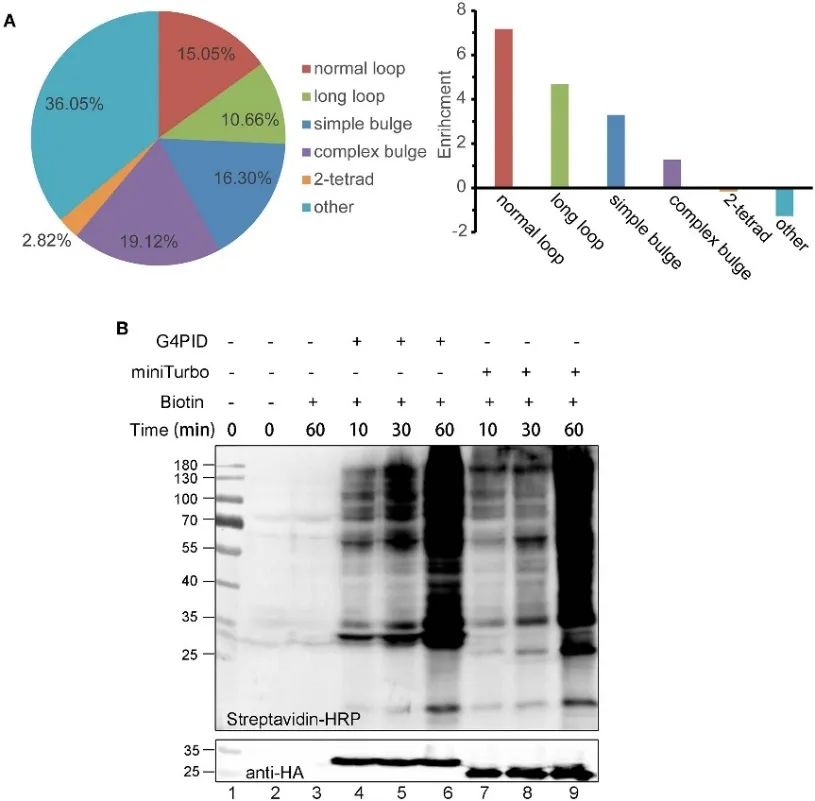
Figure 2 G 4 PID combiné avec des expériences sur l'ARN G4 et une optimisation de l'efficacité du marquage dans les cellules HEK293T.
3. Capturer et détecter le protéome d’interaction intracellulaire G4 à l’aide de la méthode PLGPB.
Par la suite, les auteurs ont utilisé la sonde G4PID pour capturer et détecter le protéome interagissant avec le G4 dans les cellules vivantes en utilisant l’approche de marquage de proximité des protéines interagissant avec le G4 (PLGPB). Dans trois expériences répétées indépendantes portant sur 578 protéines, atteignant une identité de 80 %, une transfection avec G4PID et miniTurbo dans des cellules HEK293T a été réalisée. Le criblage a révélé 149 protéines candidates interagissant avec G4, présentant un chevauchement significatif avec les bases de données existantes de protéines interagissant avec G4. En outre, l’analyse d’enrichissement de Gene Ontology (GO) a indiqué une association substantielle de ces protéines avec la régulation transcriptionnelle, l’épissage de l’ARNm et le remodelage de la chromatine, ce qui est cohérent avec les résultats antérieurs de la recherche G4.
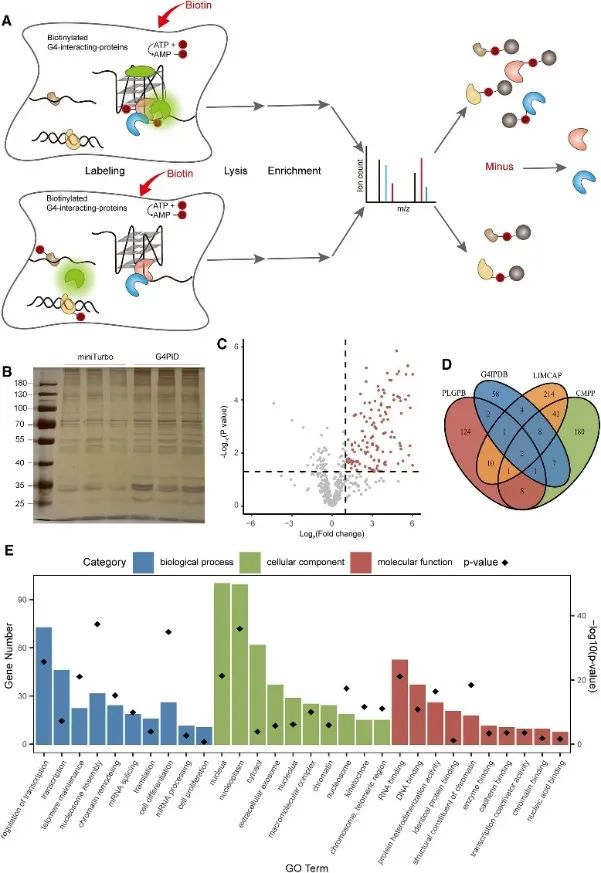
La figure 3 a identifié les protéines interagissant avec G4 par la méthode PLGPB.
4. Validation de l'affinité de liaison G4 chez les candidats nouvellement identifiés.
Grâce à la méthode PLGPB, les auteurs ont découvert une gamme de candidats englobant diverses catégories fonctionnelles, notamment des protéines communes associées aux télomères, des facteurs de transcription et des protéines de liaison aux protéines relativement rares. Cela indique que la méthode PLGPB non seulement réaffirme les protéines interagissant avec le G4 précédemment reconnues, mais élargit également de manière significative le paysage protéique en découvrant de nouveaux candidats couvrant diverses classes fonctionnelles. De plus, les auteurs ont validé la capacité de liaison de ces candidats à l’ARN G4 et à l’ADN G4, révélant une préférence prononcée pour l’ARN G4 et des variations d’affinité envers l’ADN G4 dans différentes conformations. Ces résultats soulignent le potentiel de moduler l’interaction protéine-G4 en régulant la conformation G4.
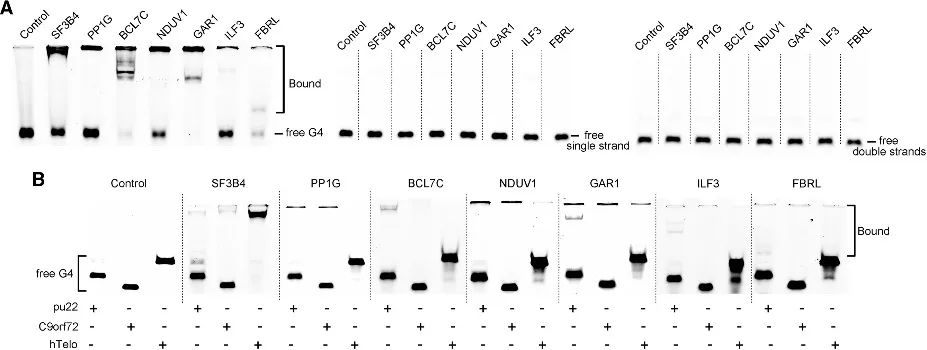
Figure 4 Interaction entre les protéines candidates et l'ARN G4 et l'ADN G4.
5. Détermination des sites de liaison à haute confiance de la structure SF3B4 et G4.
Au cours de l’étude du mode de liaison in vivo du G4, les auteurs ont mis en évidence le rôle important de la protéine SF3B4. SF3B4, connu pour son implication dans l'épissage de l'ARN, a vu ses sites de liaison à haut niveau de confiance identifiés grâce à des expériences eCLIP et une analyse complète des données rG4-seq et DMS-seq. Environ la moitié de ces sites de liaison contenaient du PQS, principalement associé à des structures G4 non conventionnelles. De plus, les auteurs ont observé une relation spatiale étroite entre les sites de liaison de SF3B4 et l'ARN PQS, avec une distribution enrichie principalement dans les régions introniques, en particulier à proximité du site d'épissage 3'. Ces résultats suggèrent fortement que l'interaction entre SF3B4 et la structure G4 pourrait jouer un rôle crucial dans les mécanismes d'épissage alternatifs.
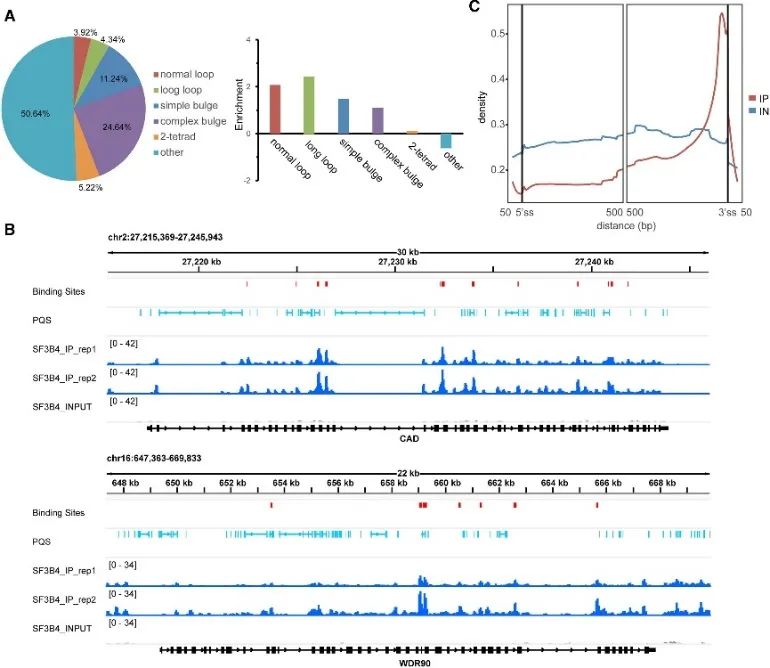
La figure 5 identifie la préférence de liaison G4 de SF3B4 dans les cellules utilisant la technologie eCLIP.
6. La structure G4 régule l'épissage alternatif médié par SF3B4.
Les auteurs ont mené une analyse des sites de liaison de SF3B4 pour dévoiler son association avec la structure G4 et ont étudié plus en détail son mécanisme de régulation dans des événements d'épissage alternatifs. Les PQS conventionnels impliqués dans l'épissage alternatif ont été criblés, avec deux gènes, INPPL1 et PPP6R2, sélectionnés pour un examen détaillé. Les structures G4 au sein de INPPL1 et PPP6R2, appelées G4-INPPL1 et G4-PPP6R2, ont été confirmées et SF3B4 a été identifié comme se liant spécifiquement à ces structures G4. En outre, il a été démontré que l’ajout du ligand G4, la pyridostatine (PDS), diminue la liaison de SF3B4 aux structures G4, favorisant ainsi les événements d’inclusion d’exons lors de l’épissage des ARNm d’INPPL1 et PPP6R2. Ces résultats indiquent l'importance potentielle de la structure G4 dans l'épissage alternatif médié par SF3B4.
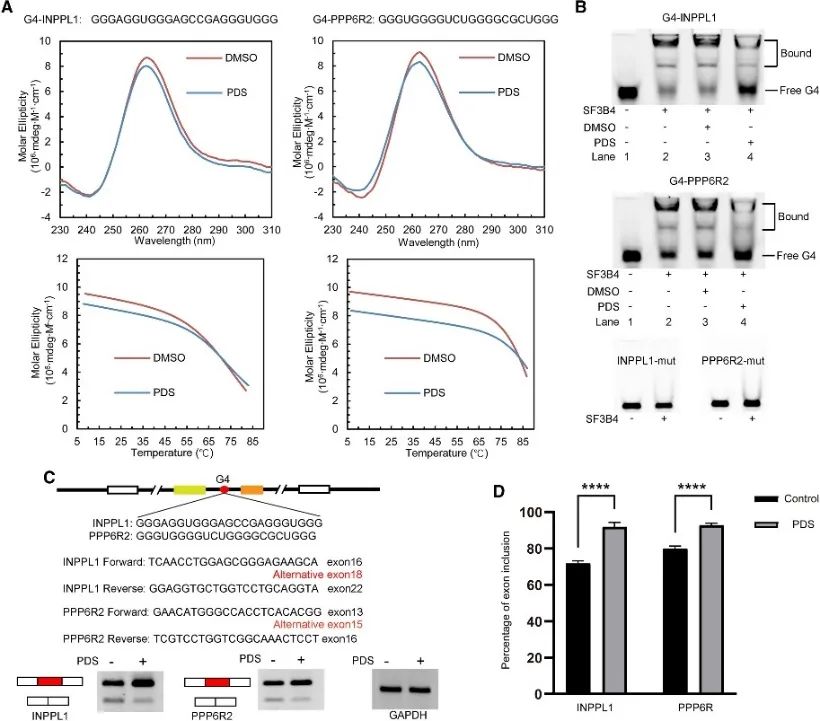
Figure 6 SF3B4 lie la structure G4 et affecte l'épissage alternatif de l'ARNm.
En conclusion, cette étude présente une boîte à outils efficace comprenant la sonde G4PID et la stratégie PLGPB, mettant en lumière la façon dont l’interaction entre G4 et les protéines au sein de la cellule influe sur l’expression des gènes, influençant particulièrement le processus crucial d’épissage alternatif. Ces résultats approfondissent notre compréhension des mécanismes de régulation impliquant le G4 dans les processus biologiques cellulaires et ouvrent la voie à une exploration plus approfondie des stratégies thérapeutiques pour les maladies liées au G4.

