-
Identification des régions d'interaction protéine-protéine, anticorps-antigène : réticulation chimique
Identification des régions d'interaction protéine-protéine, anticorps-antigène : réticulation chimique Comprendre la structure et les interactions des protéines est essentiel pour découvrir leurs fonctions biologiques. En raison de la complexité du protéome, aucune technique ne peut à elle seule révéler pleinement ces aspects. Les biologistes utilisent souvent une combinaison de méthodes, la spectrométrie de masse à réticulation (XL-MS) se distinguant par ses avantages uniques. XL-MS fournit des informations précises sur la distance spatiale entre les acides aminés re... -

Identification de l'interaction protéine-protéine dans les cellules vivantes : étiquetage de proximité
Identification de l'interaction protéine-protéine dans les cellules vivantes : plateforme technique d'étiquetage de proximité La plateforme technologique TurboID consiste à fusionner une biotine ligase mutée à l'extrémité C-terminale d'une protéine cible. Dans les cellules surexprimées, l’ajout de biotine biotinyle rapidement les protéines en interaction en 10 minutes, même à température ambiante, avec un bruit de fond minimal. Cette polyvalence rend TurboID adapté à un large éventail de systèmes biologiques, notamment les cellules de mammifères, les pla... -
Interactions protéine-protéine
Interactions protéine-protéine Les interactions protéine-protéine (IPP) constituent la pierre angulaire des activités de la vie cellulaire, jouant un rôle crucial dans la signalisation cellulaire, l'assemblage structurel et les processus vitaux clés tels que la reconnaissance pathogène-hôte. Cependant, de nombreux processus pathologiques sont également provoqués par des anomalies des IPP. Par conséquent, l’intervention et la régulation des IPP ont montré un énorme potentiel en tant que moyen de traiter les maladies associées. ChomiX intègre une série de plateformes technologiques de pointe... -
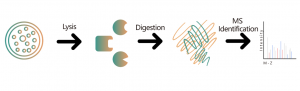
Identification des cibles et analyse de sélectivité des dégradeurs de protéines ciblés
Identification des cibles et analyse de sélectivité des dégradeurs de protéines ciblés Le médicament PROTAC (Proteolysis Targeting Chimeras) est une molécule bifonctionnelle qui peut se lier à la protéine ciblée et recruter l'ubiquitine E3 ligase pour la dégradation. Par conséquent, de nouvelles modalités telles que PROTAC et la colle moléculaire ont la capacité de modifier par inadvertance l’abondance des protéines endogènes, ce qui constitue une nouvelle méthode thérapeutique pour les cibles protéiques, en particulier les protéines non médicamentables. Globalement... -
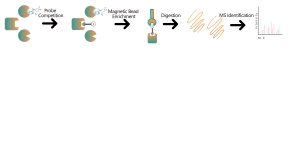
Analyse quantitative de l'occupation et de la sélectivité des cibles de médicaments covalents à petites molécules
Analyse quantitative de l'occupation et de la sélectivité des cibles de médicaments covalents à petites molécules Les médicaments covalents fonctionnent principalement en formant des liaisons covalentes avec des résidus d'acides aminés spécifiques sur des protéines cibles, telles que la cystéine, la lysine et la sérine. L’aspirine est l’une des premières molécules médicamenteuses covalentes connues. De plus, de nombreux produits naturels présentent des propriétés covalentes, comme l’oridonine, qui possède une bioactivité anti-inflammatoire. Ces dernières années, les médicaments ciblés covalents ont gagné en popularité. -
Identification du site de liaison des médicaments à petites molécules non covalentes dans les cellules vivantes
Identification du site de liaison des médicaments à petites molécules non covalentes dans les cellules vivantes. Au niveau cellulaire, l'étude directe du mode d'action entre les médicaments et les protéines cibles peut aider à éviter les changements dans la structure des protéines pendant la purification et les résultats faussement positifs qui peuvent résulter d'une réaction artificielle. systèmes tampons et concentrations élevées de médicaments et de protéines. Plateforme technique ChomiX fournit des services de protéomique chimique utilisant des sondes chimiques photoréactives biologiquement actives (avec... -
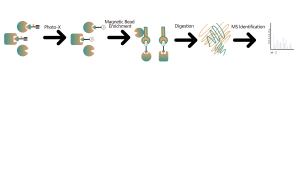
Identification de cibles médicamenteuses à petites molécules non covalentes basée sur des sondes de photoaffinité
Identification de cibles médicamenteuses à petites molécules non covalentes basée sur des sondes de photoaffinité Plateforme technique La plateforme d'identification de cibles de protéomique chimique basée sur des sondes de photoaffinité implique plusieurs étapes clés, notamment la conception de sondes, la synthèse, l'évaluation de l'activité, le marquage, l'enrichissement en protéines et l'analyse des données. Les médicaments à petites molécules non covalentes, tels que les composés synthétiques, les extraits de plantes, les produits naturels et les métabolites, peuvent être modifiés en sondes de photoaffinité. Sur... -
Interactions petites molécules-protéines
Interactions petites molécules-protéines Les protéines, en tant que participants directs et exécutants des activités de la vie, représentent des cibles cruciales pour le traitement des maladies. Les médicaments à petites molécules (composés organiques généralement d’un poids moléculaire inférieur à 1 000 Da) exercent des effets thérapeutiques efficaces en modulant finement les activités, les abondances et les interactions des protéines. Les médicaments courants à petites molécules comprennent les produits naturels et leurs dérivés (par exemple, les monomères à base de plantes) ainsi que les médicaments synthétisés chimiquement. Sur... -
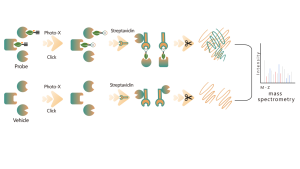
Plateforme de découverte Target : Démêler les mécanismes des produits naturels
Le uningrédient actifdansLa médecine chinoise est unesérie decomposésça haveactivité thérapeutique ou physiologiqueies. Les médicaments chinois sont riches en variété, de composition complexe et contiennent une large gamme d'ingrédients actifs,constituantun importantpossibilité d'acquériringrédients actifs, composés de plomb et makingmédicaments.
Notreplateforme de chimioprotéomiqueexcelle dansdécouvriringcibles protéiquespour nproduits naturels en médecine chinoise. C'est ingénieuxtransformerc'est çaproduits naturels en sondes chimiques multifonctionnelles, reflétant leur biographieactivités.Ces sondes, lorsqu'elles sont appliquées à des cellules vivantes ou à des tissus malades, peuventcapturer directementprotéines naturelles liant les produits. À l'aide de réactions de couplage bioorthogonales, nous isolons et enrichissons précisément ces protéines cibles. Utiliserspectre de masse haute résolutionométrie, nous obtenons une précision extrême jusque dans les pochettes de reliure. Il nous équipe dedes informations plus complètes et précises, prêt à dévoilerlecomplexemécanismesous-tendant leingrédients actifs de la médecine chinoise.
-
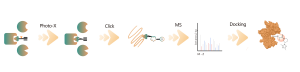
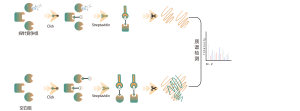
Identification de poches de liaison non covalentes pour petites molécules dans les cellules
Identification de poches de liaison non covalentes à petites molécules dans les cellules Caractéristiques techniques de la plate-forme Il est essentiel de déterminer le mode de liaison entre les médicaments à petites molécules et leurs cibles protéiques pour la R&D de médicaments. Une analyse complète de ces interactions aux niveaux structurel et physico-chimique pourrait approfondir considérablement notre compréhension des fonctions des protéines et faciliter la conception et l’optimisation des médicaments. Techniques de biologie structurale, notamment radiographie, cryo-microscopie électronique... -
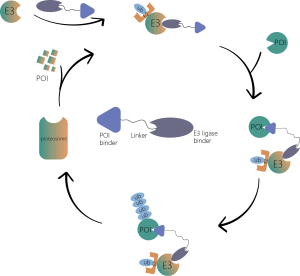
Profilage à l’échelle du protéome pour les médicaments contre la dégradation ciblée des protéines (TPD)
Le médicament PROTAC (Proteolysis Targeting Chimeras) est une molécule bifonctionnelle qui peut se lier à la protéine ciblée et recruter l’ubiquitine E3 ligase pour la dégradation. Par conséquent, de nouvelles modalités telles que PROTAC et la colle moléculaire ont la capacité de modifier par inadvertance l’abondance des protéines endogènes, ce qui constitue une nouvelle méthode thérapeutique pour les cibles protéiques, en particulier les protéines non médicamentables. La quantification complète de l’abondance des protéines cibles et non ciblées a été l’une des expériences standards pour la R&D sur les médicaments TPD.
-
Identification de cibles protéiques par protéomique différentielle
Identification de cibles protéiques par plate-forme de protéomique différentielle Caractéristiques techniques La protéomique différentielle étudie les modifications du protéome sous différents états physiologiques ou pathologiques, tels que les traitements médicamenteux ou la régulation génique, en comparant deux échantillons ou plus. Cette approche met en lumière des processus vitaux importants ou des maladies majeures afin de déterminer les différentes protéines clés considérées comme des marqueurs pour l'analyse qualitative et fonctionnelle. Des milliers de protéines peuvent être identifiées...

