Interazioni acido nucleico-proteine rivelate attraverso la strategia di etichettatura di prossimità: esplorazione delle sonde G4PID e della strategia PLGPB
Questo articolo presenta uno studio innovativo in cui gli autori introducono una nuova sonda bifunzionale denominata G4PID. Questa sonda combina il dominio di legame G4 (RHAU23) di RHAU con la miniTurbo biotina ligasi, consentendo il targeting preciso dei G-quadruplex (G4) e l'etichettatura delle proteine che interagiscono con G4 all'interno delle cellule vive. Le strutture G4, formate da sequenze di acidi nucleici ricchi di guanina, sono fondamentali in vari processi cellulari tra cui la regolazione genetica, la replicazione del DNA e la riparazione. Nonostante l’ampio riconoscimento dell’importanza di G4, i meccanismi specifici all’interno delle cellule e le loro interazioni con le proteine rimangono in gran parte inesplorati. Utilizzando G4PID, gli autori hanno sviluppato un metodo specializzato di etichettatura della biotina (PLGPB) per identificare e analizzare accuratamente le proteine che interagiscono con G4, facendo luce sul ruolo cruciale delle strutture G4 nelle funzioni cellulari. Questo approccio ha identificato con successo 149 proteine candidate che interagiscono con G4, prevalentemente coinvolte nella regolazione trascrizionale, nello splicing dell'mRNA e nel rimodellamento della cromatina. La validazione di sette proteine candidate ha rivelato la loro preferenza per il legame dell'RNA G4, anche se con affinità variabili verso il DNA G4. L'esame dettagliato del fattore di splicing SF3B4 ha dimostrato la sua interazione con le strutture G4, influenzando gli eventi di splicing alternativi. La stabilizzazione delle strutture G4 ha alterato l'efficienza di legame di SF3B4 e ha influenzato lo splicing alternativo di geni specifici (ad esempio, INPPL1 e PPP6R2).

La berberina, un alcaloide tradizionale con effetti farmacologici ad ampio raggio tra cui antinfiammatori, ipoglicemia e protezione cardiovascolare, ha attirato notevole attenzione. Tuttavia, i suoi precisi meccanismi molecolari, in particolare nella soppressione dell’infiammazione, rimangono poco chiari.
Questo studio colma questa lacuna di conoscenza utilizzando la tecnologia ABPP per identificare EIF2AK2 come un bersaglio critico utilizzato dalla berberina per la sua azione antinfiammatoria. I risultati approfondiscono la nostra comprensione del meccanismo della berberina e forniscono una base scientifica per riposizionare la berberina e sviluppare nuovi farmaci antinfiammatori mirati a EIF2AK2.
Il team ha utilizzato metodi chemioproteomici avanzati per studiare sistematicamente le interazioni della berberina con le proteine intracellulari, confermando il suo legame specifico con EIF2AK2 e la modulazione della sua attività enzimatica. Ciò influenza i percorsi di risposta infiammatoria, inibendo efficacemente la progressione dell’infiammazione. Questa significativa innovazione offre approfondimenti sul meccanismo antinfiammatorio della berberina e supporta lo sviluppo di nuove terapie mirate a EIF2AK2.
ChomiX fornisce servizi all'avanguardia come ABPP e CETSA per aiutare i ricercatori a esplorare i meccanismi dei farmaci e ad accelerare gli sforzi di sviluppo di nuovi farmaci.
Itinerario di ricerca

Processo sperimentale
1. Verifica della costruzione e della specificità della sonda G4PID.
Gli autori inizialmente hanno progettato la sonda G4PID, con il dominio di legame G4 RHAU23 posizionato all'estremità N e il miniTurbo all'estremità C, collegati da un linker flessibile. L'espressione e la purificazione sono state effettuate utilizzando il ceppo BL21 (DE3). Esperimenti specifici di validazione del legame con G4PID purificato hanno rivelato il suo preciso legame con BCL2 G4, esibendo un valore Kd di 15 ± 7 nM, mentre mostrava un'affinità trascurabile verso altre strutture non G4. Inoltre, gli autori hanno valutato la specificità della biotinilazione di G4PID, dimostrando la sua capacità di biotinilare selettivamente le proteine che interagiscono con G4.
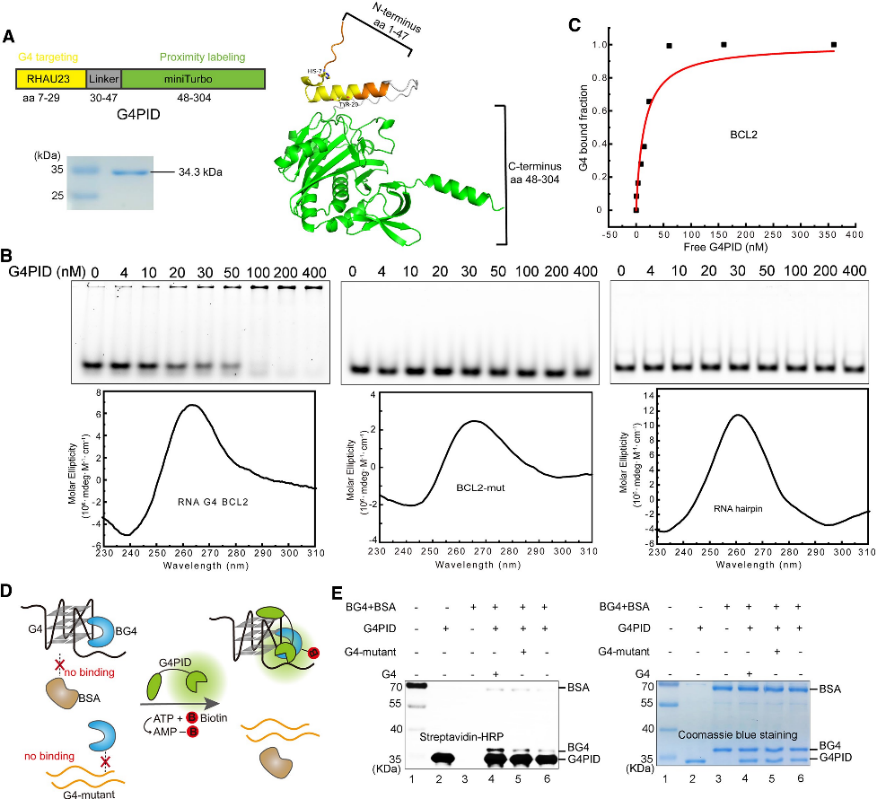
Figura 1 Il PID G4 media la biotinilazione specifica della proteina che interagisce con G4
2. Capacità di legame dell'RNA G4 di G4PID nelle cellule e caratteristiche di etichettatura della biotina.
Gli autori hanno creato linee cellulari HA-G4PID e HA-miniTurbo HeLa per studiare l'affinità di legame di G4PID con l'RNA G4 intracellulare tramite esperimenti eCLIP. I risultati dimostrano l'interazione selettiva di G4PID con RNA G4, esibendo un'affinità di legame notevolmente più elevata per le sequenze di RNA contenenti PQS rispetto alle sequenze casuali. Inoltre, gli autori hanno osservato caratteristiche uniche di G4PID che facilitano l'inizio della marcatura della biotina all'interno delle cellule senza richiedere cofattori. Ciò ha portato a un miglioramento significativo dell’efficienza di etichettatura delle proteine, con evidenti segnali di biotinilazione rilevati per G4PID entro 30 minuti, indicando una cinetica di etichettatura più rapida rispetto a miniTurbo.
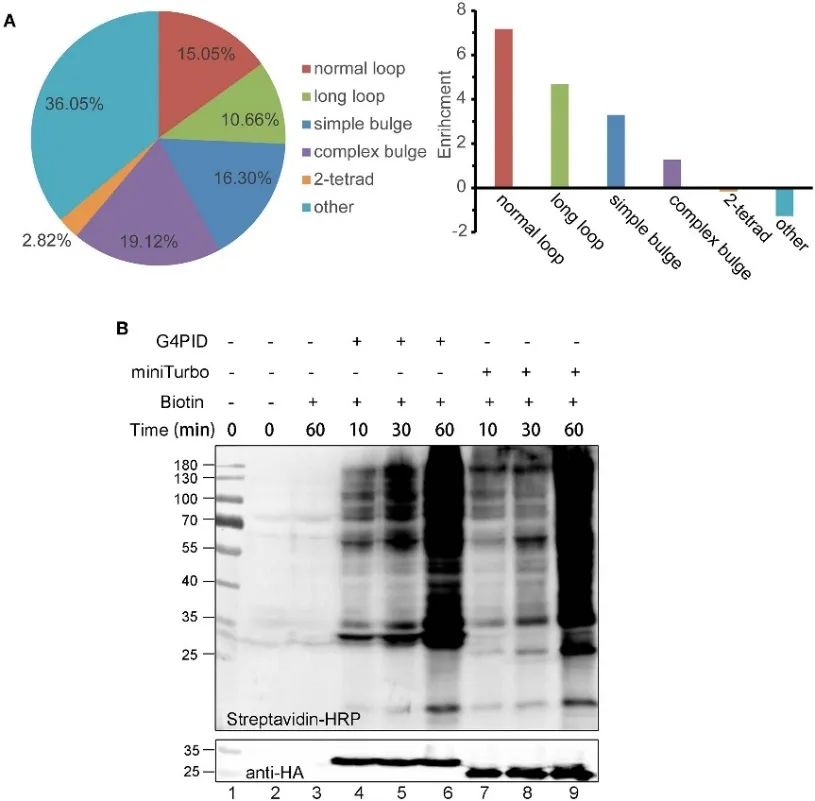
Figura 2 PID G 4 combinato con esperimenti di RNA G4 e ottimizzazione per l'efficienza di etichettatura nelle cellule HEK293T.
3. Catturare e rilevare il proteoma interagente G4 intracellulare utilizzando il metodo PLGPB.
Successivamente, gli autori hanno utilizzato la sonda G4PID per catturare e rilevare il proteoma interagente con G4 all'interno di cellule vive utilizzando l'approccio Proximity Labeling of G4-Interacting Proteins (PLGPB). In tre esperimenti replicati indipendenti comprendenti 578 proteine, raggiungendo un'identità dell'80%, è stata condotta la trasfezione con G4PID e miniTurbo nelle cellule HEK293T. Lo screening ha rivelato 149 proteine candidate che interagiscono con G4, mostrando una significativa sovrapposizione con i database esistenti di proteine che interagiscono con G4. Inoltre, l'analisi di arricchimento di Gene Ontology (GO) ha indicato un'associazione sostanziale di queste proteine con la regolazione trascrizionale, lo splicing dell'mRNA e il rimodellamento della cromatina, in linea con i risultati precedenti della ricerca G4.
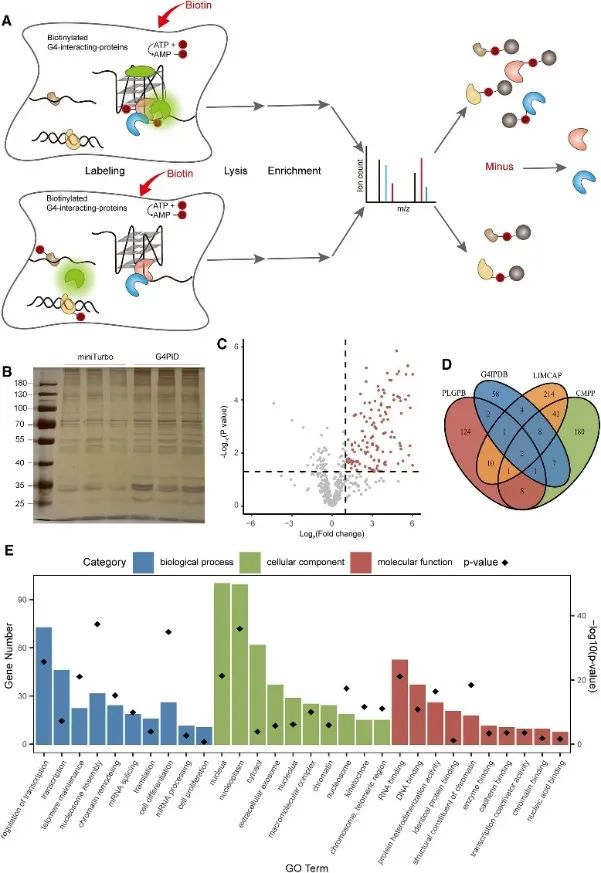
La Figura 3 ha identificato le proteine che interagiscono con G4 mediante il metodo PLGPB.
4. Convalida dell'affinità di legame G4 nei candidati appena identificati.
Attraverso il metodo PLGPB, gli autori hanno scoperto una serie di candidati che comprendono varie categorie funzionali, tra cui proteine comuni associate ai telomeri, fattori di trascrizione e proteine leganti le proteine relativamente non comuni. Ciò indica che il metodo PLGPB non solo riafferma le proteine interagenti G4 precedentemente riconosciute, ma amplia anche significativamente il panorama proteico scoprendo nuovi candidati che abbracciano diverse classi funzionali. Inoltre, gli autori hanno convalidato la capacità di legame di questi candidati all'RNA G4 e al DNA G4, rivelando una preferenza pronunciata per l'RNA G4 e variazioni di affinità verso il DNA G4 in diverse conformazioni. Questi risultati sottolineano il potenziale di modulare l'interazione proteina-G4 regolando la conformazione di G4.
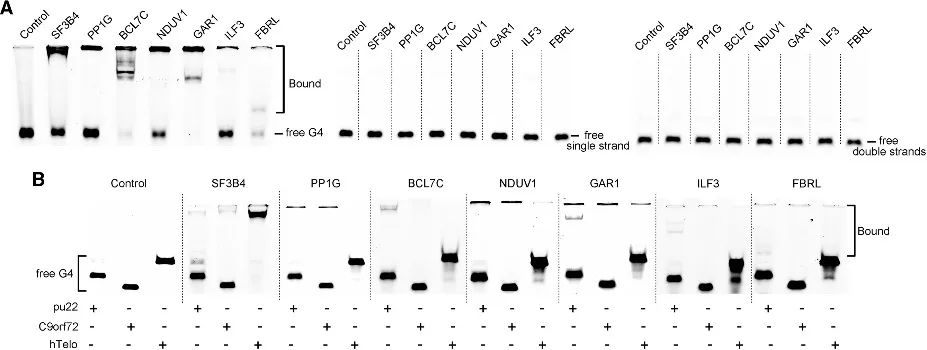
Figura 4 Interazione tra le proteine candidate e RNA G4 e DNA G4.
5. Determinazione dei siti di legame ad alta confidenza della struttura SF3B4 e G4.
Durante l'indagine sulla modalità in vivo del legame G4, gli autori hanno portato alla luce il ruolo significativo della proteina SF3B4. SF3B4, noto per il suo coinvolgimento nello splicing dell'RNA, ha identificato i suoi siti di legame ad alta sicurezza attraverso esperimenti eCLIP e un'analisi completa dei dati rG4-seq e DMS-seq. Circa la metà di questi siti di legame contenevano PQS, prevalentemente associati a strutture G4 non convenzionali. Inoltre, gli autori hanno osservato una stretta relazione spaziale tra i siti di legame di SF3B4 e l'RNA PQS, con una distribuzione arricchita principalmente all'interno delle regioni introniche, in particolare prossimali al sito di giunzione 3'. Questi risultati suggeriscono fortemente che l'interazione tra SF3B4 e la struttura G4 potrebbe svolgere un ruolo cruciale nei meccanismi di splicing alternativo.
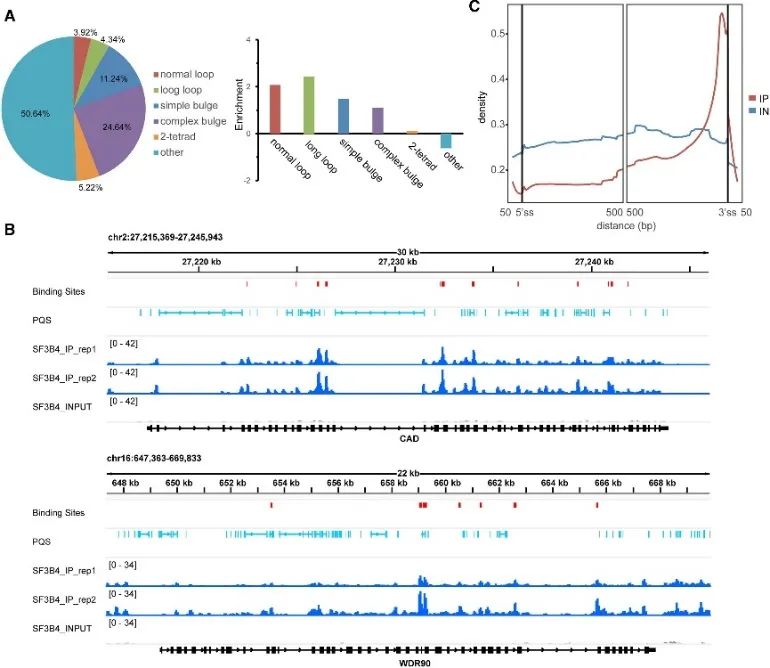
La Figura 5 identifica la preferenza di legame G4 di SF3B4 nelle cellule che utilizzano la tecnologia eCLIP.
6. La struttura G4 regola lo splicing alternativo mediato da SF3B4.
Gli autori hanno condotto un'analisi dei siti di legame di SF3B4 per svelare la sua associazione con la struttura G4 e hanno studiato ulteriormente il suo meccanismo di regolazione negli eventi di splicing alternativo. Sono stati esaminati i PQS convenzionali implicati nello splicing alternativo, con due geni, INPPL1 e PPP6R2, selezionati per un esame dettagliato. Sono state confermate le strutture G4 all'interno di INPPL1 e PPP6R2, denominate G4-INPPL1 e G4-PPP6R2, e SF3B4 è stato identificato come specifico legame con queste strutture G4. Inoltre, è stato dimostrato che l'aggiunta del ligando G4 piridostatina (PDS) diminuisce il legame di SF3B4 alle strutture G4, promuovendo di conseguenza eventi di inclusione dell'esone durante lo splicing dell'mRNA di INPPL1 e PPP6R2. Questi risultati indicano il potenziale significato della struttura G4 nello splicing alternativo mediato da SF3B4.
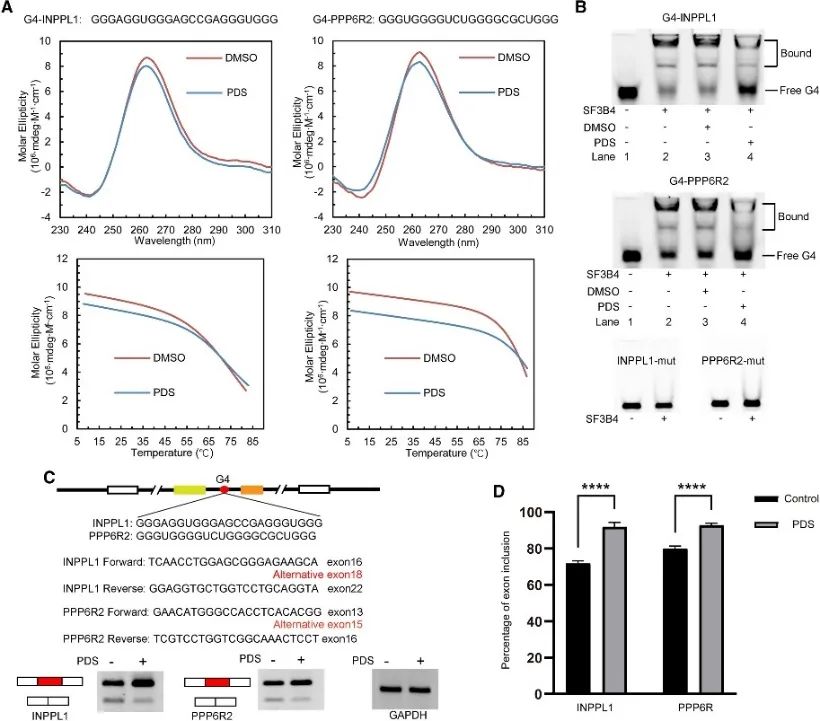
Figura 6 SF3B4 lega la struttura G4 e influenza lo splicing alternativo dell'mRNA.
In conclusione, questo studio introduce un efficiente kit di strumenti comprendente la sonda G4PID e la strategia PLGPB, facendo luce su come l'interazione tra G4 e le proteine all'interno della cellula influisce sull'espressione genica, influenzando in particolare il processo fondamentale dello splicing alternativo. Questi risultati approfondiscono la nostra comprensione dei meccanismi regolatori che coinvolgono G4 nei processi biologici cellulari e aprono la strada a ulteriori esplorazioni nelle strategie terapeutiche per le malattie correlate a G4.

