근접 라벨링 전략을 통해 밝혀진 핵산-단백질 상호 작용: G4PID 프로브 및 PLGPB 전략 탐색
이 기사는 저자가 G4PID라는 새로운 이중 기능 프로브를 소개하는 혁신적인 연구를 제시합니다. 이 프로브는 RHAU의 G4 결합 도메인(RHAU23)과 miniTurbo 비오틴 리가제를 결합하여 G-quadruplexes(G4)의 정확한 타겟팅을 가능하게 하고 살아있는 세포 내에서 G4 상호 작용 단백질에 태그를 지정할 수 있습니다. 구아닌이 풍부한 핵산 서열로 형성된 G4 구조는 유전자 조절, DNA 복제 및 복구를 포함한 다양한 세포 과정에서 중추적인 역할을 합니다. G4의 중요성에 대한 광범위한 인식에도 불구하고, 세포 내의 특정 메커니즘과 단백질과의 상호 작용은 아직 대부분 탐구되지 않은 상태로 남아 있습니다. 저자는 G4PID를 사용하여 G4 상호작용 단백질을 정확하게 식별하고 분석하기 위한 특수 비오틴 라벨링 방법(PLGPB)을 개발하여 세포 기능에서 G4 구조의 중요한 역할을 밝혀냈습니다. 이 접근법은 주로 전사 조절, mRNA 접합 및 염색질 리모델링에 관여하는 G4와 상호 작용하는 149개의 단백질 후보를 성공적으로 식별했습니다. 7가지 후보 단백질의 검증을 통해 DNA G4에 대한 친화력은 다양하지만 RNA G4 결합에 대한 선호도가 밝혀졌습니다. 접합 인자 SF3B4에 대한 자세한 조사에서는 G4 구조와의 상호 작용이 입증되어 대체 접합 이벤트에 영향을 미쳤습니다. G4 구조의 안정화는 SF3B4의 결합 효율을 변경하고 특정 유전자(예: INPPL1 및 PPP6R2)의 대체 접합에 영향을 미쳤습니다.

항염증, 저혈당증, 심혈관 보호 등 광범위한 약리 효과를 지닌 전통적인 알칼로이드인 베르베린(Berberine)이 상당한 주목을 받고 있습니다. 그러나 특히 염증 억제에서의 정확한 분자 메커니즘은 아직 불분명합니다.
이 연구는 EIF2AK2를 베르베린의 항염증 작용에 관여하는 중요한 표적으로 식별하기 위해 ABPP 기술을 사용하여 이러한 지식 격차를 메웁니다. 이번 발견은 베르베린의 메커니즘에 대한 이해를 심화시키고 베르베린의 위치를 재설정하고 새로운 EIF2AK2 표적 항염증제를 개발하기 위한 과학적 기반을 제공합니다.
연구팀은 베르베린과 세포내 단백질의 상호작용을 체계적으로 조사하기 위해 고급 화학단백체 방법을 사용하여 EIF2AK2에 대한 특이적인 결합과 효소 활성의 조절을 확인했습니다. 이는 염증 반응 경로에 영향을 주어 염증 진행을 효과적으로 억제합니다. 이 중요한 혁신은 베르베린의 항염증 메커니즘에 대한 통찰력을 제공하고 EIF2AK2를 표적으로 하는 새로운 치료법 개발을 지원합니다.
ChomiX는 연구자들이 약물 메커니즘을 탐색하고 신약 개발 노력을 가속화할 수 있도록 ABPP 및 CETSA와 같은 최첨단 서비스를 제공합니다.
연구 경로

실험 과정
1. G4PID 프로브의 구성 및 특이성 검증.
저자는 처음에 G4 결합 도메인 RHAU23이 N 말단에 위치하고 miniTurbo가 C 말단에 위치하며 유연한 링커로 연결된 G4PID 프로브를 설계했습니다. 발현 및 정제는 BL21(DE3) 균주를 사용하여 수행하였다. 정제된 G4PID를 사용한 특이적 결합 검증 실험에서는 BCL2 G4에 대한 정확한 결합이 밝혀졌으며 15 ± 7 nM의 Kd 값을 나타냈지만 다른 비-G4 구조에 대해서는 무시할 수 있는 친화력을 나타냈습니다. 또한, 저자는 G4PID의 비오티닐화 특이성을 평가하여 G4 상호작용 단백질을 선택적으로 비오티닐화하는 능력을 입증했습니다.
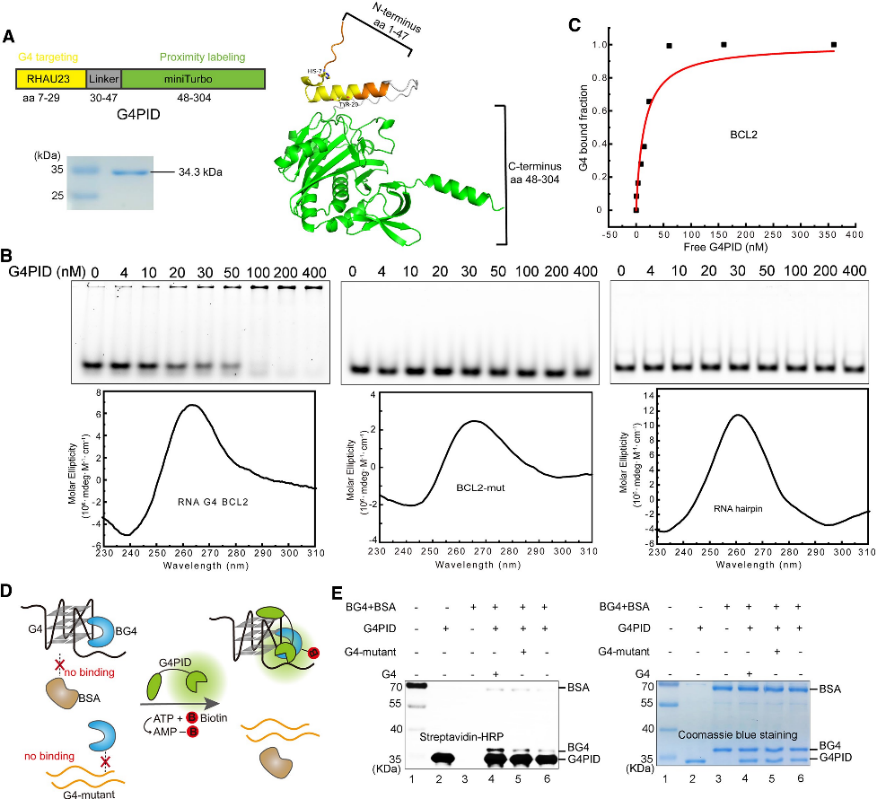
그림 1 G4 PID는 G4 상호작용 단백질의 특정 비오티닐화를 매개합니다.
2. 세포 내 G4PID의 RNA G4 결합 능력 및 비오틴 표지 특성.
저자는 eCLIP 실험을 통해 세포내 RNA G4에 대한 G4PID의 결합 친화성을 조사하기 위해 HA-G4PID 및 HA-miniTurbo HeLa 세포주를 확립했습니다. 결과는 G4PID와 RNA G4의 선택적 상호 작용을 보여 주며, 무작위 서열에 비해 PQS를 포함하는 RNA 서열에 대해 현저히 높은 결합 친화력을 나타냅니다. 또한 저자는 보조 인자 없이도 세포 내에서 비오틴 표지의 시작을 촉진하는 G4PID의 독특한 특징을 관찰했습니다. 이로 인해 단백질 라벨링 효율성이 크게 향상되었으며, 30분 이내에 G4PID에 대한 확실한 비오티닐화 신호가 감지되어 miniTurbo에 비해 라벨링 동역학이 더 빠르다는 것을 알 수 있습니다.
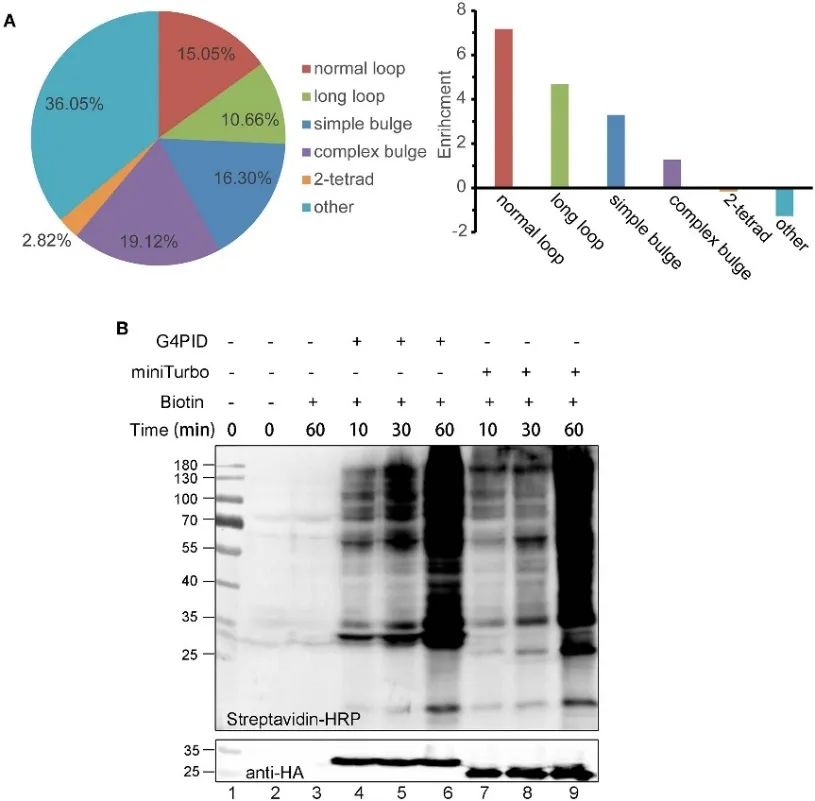
그림 2 G 4 PID와 RNA G4 실험 및 HEK293T 세포의 라벨링 효율성 최적화가 결합되었습니다.
3. PLGPB 방법을 사용하여 세포내 G4 상호 작용 프로테옴을 캡처하고 감지합니다.
그 후, 저자는 G4 상호작용 단백질의 근접 라벨링(PLGPB) 접근 방식을 사용하여 살아있는 세포 내에서 G4 상호 작용 프로테옴을 캡처하고 감지하기 위해 G4PID 프로브를 사용했습니다. 578개의 단백질을 포함하는 3개의 독립적인 반복 실험에서 80% 동일성을 달성하고 HEK293T 세포에서 G4PID 및 miniTurbo를 사용한 형질감염을 수행했습니다. 스크리닝 결과 G4와 상호작용하는 149개의 후보 단백질이 밝혀졌으며, 이는 G4 상호작용 단백질의 기존 데이터베이스와 상당한 중복을 나타냈습니다. 또한, Gene Ontology(GO) 농축 분석은 G4 연구의 이전 연구 결과와 일치하는 전사 조절, mRNA 접합 및 염색질 리모델링과 이들 단백질의 실질적인 연관성을 나타냅니다.
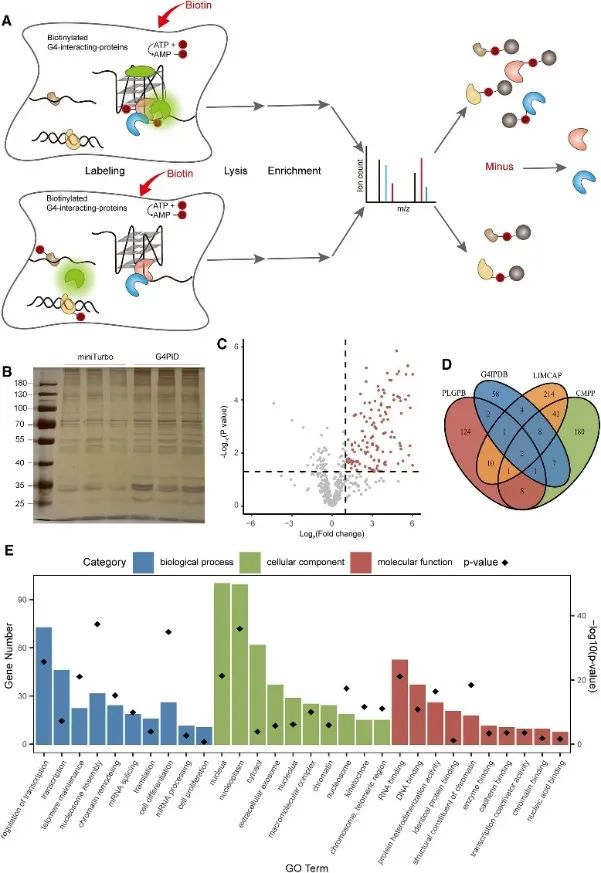
그림 3은 PLGPB 방법으로 G4와 상호 작용하는 단백질을 확인했습니다.
4. 새로 식별된 후보의 G4 결합 친화도 검증.
PLGPB 방법을 통해 저자는 일반적인 텔로미어 관련 단백질, 전사 인자 및 비교적 흔하지 않은 단백질 결합 단백질을 포함하여 다양한 기능 범주를 포괄하는 다양한 후보를 발견했습니다. 이는 PLGPB 방법이 이전에 인정된 G4 상호 작용 단백질을 재확인할 뿐만 아니라 다양한 기능 클래스에 걸친 새로운 후보를 밝혀 단백질 환경을 크게 넓힌다는 것을 나타냅니다. 더욱이, 저자들은 이들 후보물질의 RNA G4 및 DNA G4에 대한 결합 능력을 검증하여 RNA G4에 대한 뚜렷한 선호도와 다양한 형태의 DNA G4에 대한 친화력의 변화를 밝혔습니다. 이러한 발견은 G4 형태를 조절함으로써 단백질-G4 상호작용을 조절할 수 있는 가능성을 강조합니다.
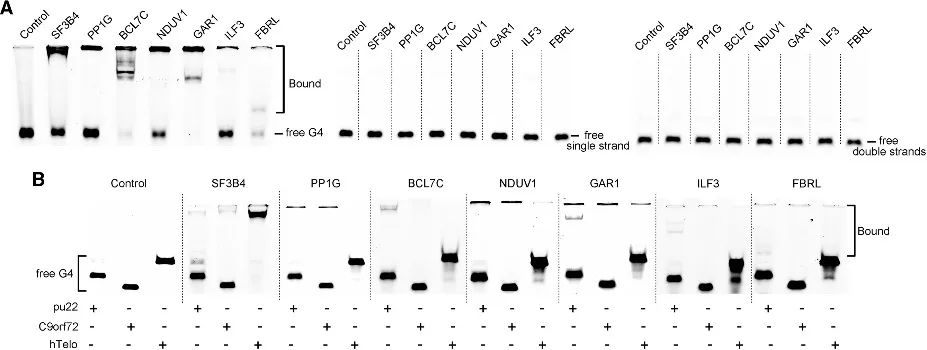
그림 4 후보 단백질과 RNA G4 및 DNA G4 간의 상호 작용.
5. SF3B4 및 G4 구조의 고신뢰성 결합 부위 결정.
G4 결합의 생체 내 모드를 조사하는 동안 저자는 SF3B4 단백질의 중요한 역할을 밝혀냈습니다. RNA 스플라이싱에 관여하는 것으로 알려진 SF3B4는 eCLIP 실험과 rG4-seq 및 DMS-seq 데이터에 대한 포괄적인 분석을 통해 확인된 높은 신뢰도의 결합 사이트를 가지고 있었습니다. 이러한 결합 부위의 약 절반에는 주로 비전통적인 G4 구조와 관련된 PQS가 포함되어 있습니다. 더욱이, 저자는 주로 인트론 영역, 특히 3' 스플라이스 부위에 근접한 분포가 풍부한 SF3B4 결합 부위와 RNA PQS 사이의 밀접한 공간적 관계를 관찰했습니다. 이러한 발견은 SF3B4와 G4 구조 사이의 상호 작용이 대체 접합 메커니즘에서 중요한 역할을 할 수 있음을 강력히 시사합니다.
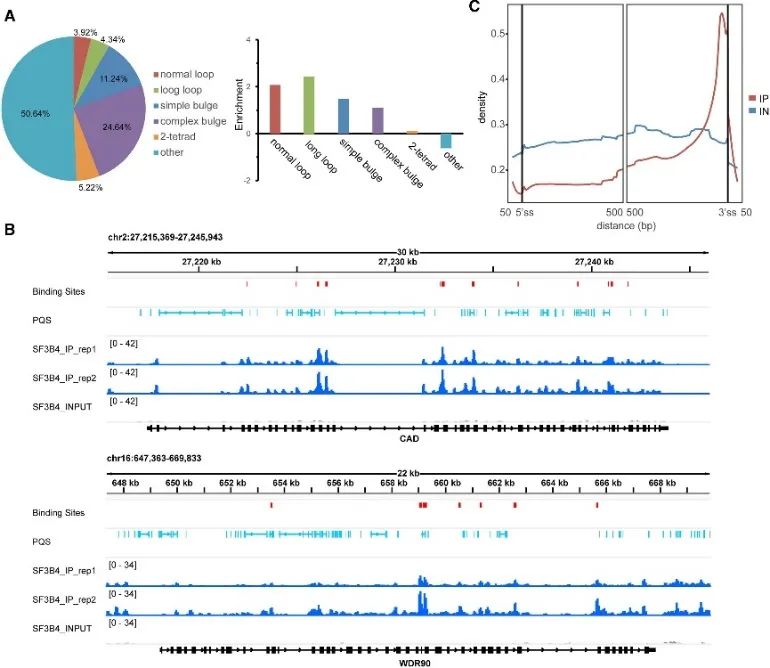
그림 5는 eCLIP 기술을 사용하여 세포에서 SF3B4의 G4 결합 선호도를 식별합니다.
6. G4 구조는 SF3B4 매개 대체 접합을 조절합니다.
저자는 G4 구조와의 연관성을 밝히기 위해 SF3B4 결합 부위 분석을 수행했으며 대체 접합 이벤트에서 SF3B4의 조절 메커니즘을 추가로 조사했습니다. 대체 스플라이싱과 관련된 기존 PQS를 스크리닝했으며, 자세한 조사를 위해 선택된 두 개의 유전자 INPPL1 및 PPP6R2를 사용했습니다. G4-INPPL1 및 G4-PPP6R2라고 불리는 INPPL1 및 PPP6R2 내의 G4 구조가 확인되었으며 SF3B4는 이러한 G4 구조에 특이적으로 결합하는 것으로 확인되었습니다. 또한, G4 리간드 피리도스타틴(PDS)을 첨가하면 G4 구조에 대한 SF3B4 결합이 감소하여 결과적으로 INPPL1 및 PPP6R2 mRNA 스플라이싱 동안 엑손 포함 이벤트가 촉진된다는 것이 입증되었습니다. 이러한 발견은 SF3B4 매개 대체 접합에서 G4 구조의 잠재적 중요성을 나타냅니다.
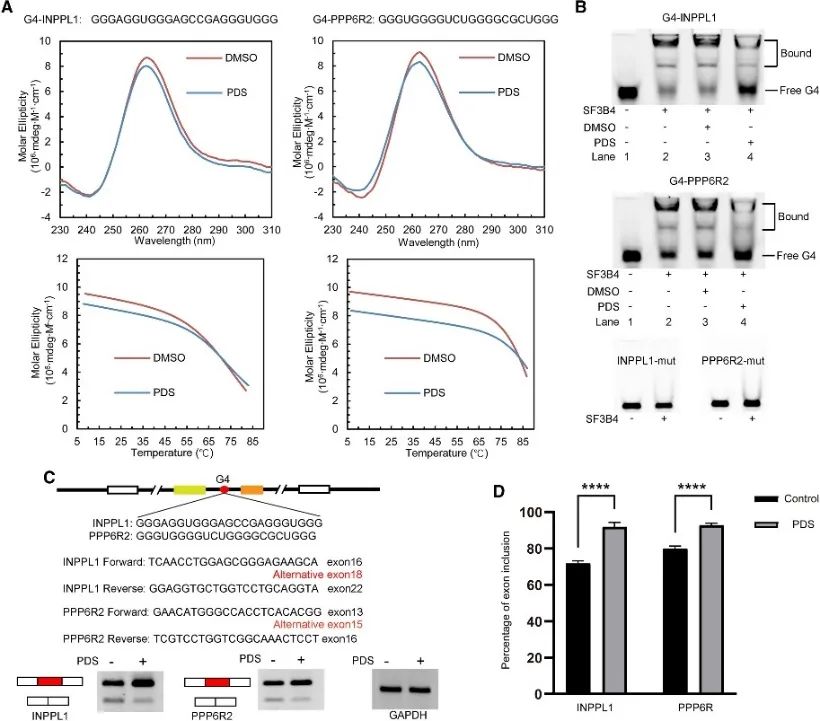
그림 6 SF3B4는 G4 구조에 결합하고 mRNA의 대체 접합에 영향을 미칩니다.
결론적으로, 이 연구는 G4PID 프로브와 PLGPB 전략으로 구성된 효율적인 툴킷을 소개하여 세포 내 G4와 단백질 간의 상호 작용이 유전자 발현에 어떻게 영향을 미치는지, 특히 대체 접합의 중추적 과정에 영향을 미치는지 밝혀줍니다. 이러한 발견은 세포 생물학적 과정에서 G4와 관련된 규제 메커니즘에 대한 이해를 심화시키고 G4 관련 질병에 대한 치료 전략에 대한 추가 탐구의 길을 열어줍니다.

