Nucleïnezuur-eiwitinteracties onthuld door middel van nabijheidslabelstrategie: onderzoek naar G4PID-sondes en PLGPB-strategie
Dit artikel presenteert een innovatief onderzoek waarin de auteurs een nieuwe bifunctionele probe introduceren, genaamd G4PID. Deze sonde combineert het G4-bindende domein (RHAU23) van RHAU met de miniTurbo-biotineligase, waardoor nauwkeurige targeting van G-quadruplexen (G4) en het taggen van G4-interagerende eiwitten in levende cellen mogelijk wordt. G4-structuren, gevormd door guaninerijke nucleïnezuursequenties, zijn cruciaal in verschillende cellulaire processen, waaronder genregulatie, DNA-replicatie en reparatie. Ondanks de wijdverbreide erkenning van de betekenis van G4, blijven de specifieke mechanismen in cellen en hun interacties met eiwitten grotendeels onontgonnen. Met behulp van G4PID ontwikkelden de auteurs een gespecialiseerde biotine-labelingsmethode (PLGPB) om G4-interagerende eiwitten nauwkeurig te identificeren en analyseren, wat licht werpt op de cruciale rol van G4-structuren in cellulaire functies. Deze aanpak identificeerde met succes 149 eiwitkandidaten die interageren met G4, voornamelijk betrokken bij transcriptionele regulatie, mRNA-splitsing en hermodellering van chromatine. Validatie van zeven kandidaat-eiwitten onthulde hun voorkeur voor RNA G4-binding, zij het met variërende affiniteiten voor DNA G4. Gedetailleerd onderzoek van de splitsingsfactor SF3B4 toonde de interactie ervan met G4-structuren aan, wat een impact had op alternatieve splitsingsgebeurtenissen. Stabilisatie van G4-structuren veranderde de bindingsefficiëntie van SF3B4 en beïnvloedde alternatieve splitsing van specifieke genen (bijv. INPPL1 en PPP6R2).

Berberine, een traditionele alkaloïde met uitgebreide farmacologische effecten, waaronder ontstekingsremmende werking, hypoglykemie en cardiovasculaire bescherming, heeft veel aandacht getrokken. De precieze moleculaire mechanismen ervan, vooral bij het onderdrukken van ontstekingen, blijven echter onduidelijk.
Deze studie vult deze kenniskloof op met behulp van ABPP-technologie om EIF2AK2 te identificeren als een cruciaal doelwit van berberine vanwege zijn ontstekingsremmende werking. De bevindingen verdiepen ons begrip van het mechanisme van berberine en bieden een wetenschappelijke basis voor het herpositioneren van berberine en het ontwikkelen van nieuwe op EIF2AK2 gerichte ontstekingsremmende medicijnen.
Het team gebruikte geavanceerde chemoproteomische methoden om de interacties van berberine met intracellulaire eiwitten systematisch te onderzoeken, waarbij de specifieke binding aan EIF2AK2 en de modulatie van de enzymatische activiteit ervan werden bevestigd. Dit beïnvloedt de ontstekingsreactieroutes, waardoor de progressie van ontstekingen effectief wordt geremd. Deze belangrijke doorbraak biedt inzicht in het ontstekingsremmende mechanisme van berberine en ondersteunt de ontwikkeling van nieuwe therapieën gericht op EIF2AK2.
ChomiX biedt geavanceerde diensten zoals ABPP en CETSA om onderzoekers te helpen bij het onderzoeken van medicijnmechanismen en het versnellen van de ontwikkeling van nieuwe medicijnen.
Onderzoeksroute

Experimenteel proces
1. Constructie en specificiteitsverificatie van de G4PID-sonde.
De auteurs ontwikkelden aanvankelijk de G4PID-sonde, met het G4-bindende domein RHAU23 gepositioneerd aan de N-terminus en de miniTurbo aan de C-terminus, verbonden door een flexibele linker. Expressie en zuivering werden uitgevoerd met behulp van de stam BL21 (DE3). Specifieke bindingsvalidatie-experimenten met gezuiverd G4PID onthulden de precieze binding ervan aan BCL2 G4, waarbij een Kd-waarde van 15 ± 7 nM werd vertoond, terwijl er een verwaarloosbare affiniteit voor andere niet-G4-structuren werd vertoond. Bovendien beoordeelden de auteurs de biotinyleringsspecificiteit van G4PID, wat het vermogen aantoont om selectief G4-interagerende eiwitten te biotinyleren.
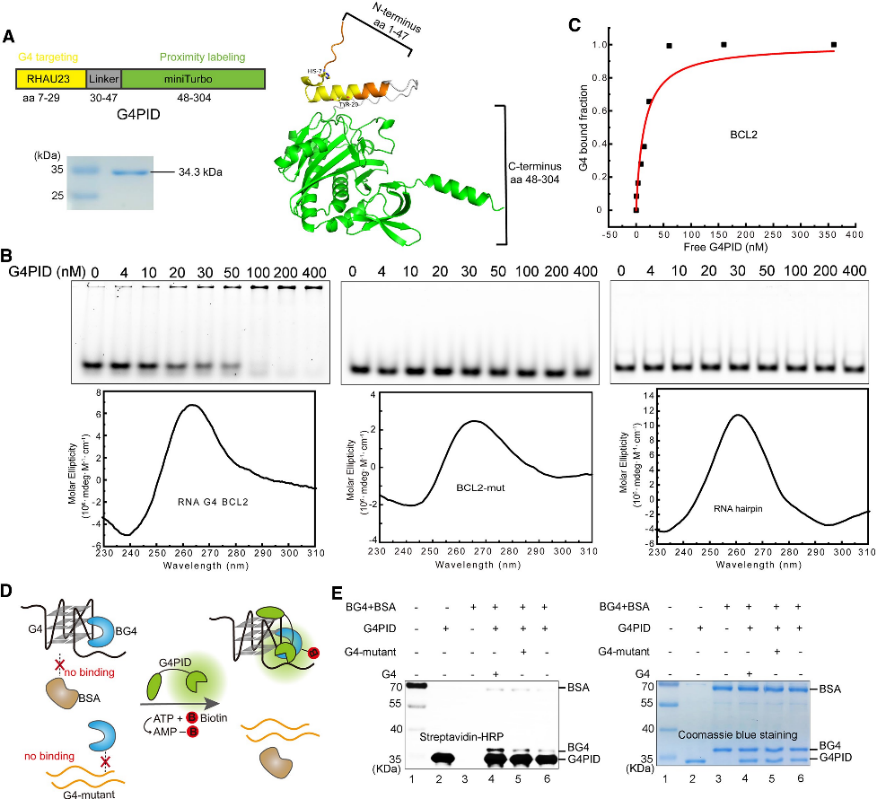
Figuur 1 G4 PID bemiddelt de specifieke biotinylatie van het G4-interagerende eiwit
2. RNA G4-bindingsvermogen van G4PID in cellen en kenmerken van biotine-labeling.
De auteurs hebben HA-G4PID- en HA-miniTurbo HeLa-cellijnen ontwikkeld om de bindingsaffiniteit van G4PID aan intracellulair RNA G4 te onderzoeken via eCLIP-experimenten. De resultaten demonstreren de selectieve interactie van G4PID met RNA G4, waarbij een opmerkelijk hogere bindingsaffiniteit voor RNA-sequenties die PQS bevatten wordt vertoond in vergelijking met willekeurige sequenties. Bovendien hebben de auteurs unieke kenmerken van G4PID waargenomen die de initiatie van biotine-labeling in cellen vergemakkelijken zonder dat cofactoren nodig zijn. Dit leidde tot een significante verbetering van de efficiëntie van eiwitlabeling, waarbij binnen 30 minuten duidelijke biotinylatiesignalen voor G4PID werden gedetecteerd, wat wijst op een snellere labelkinetiek vergeleken met miniTurbo.
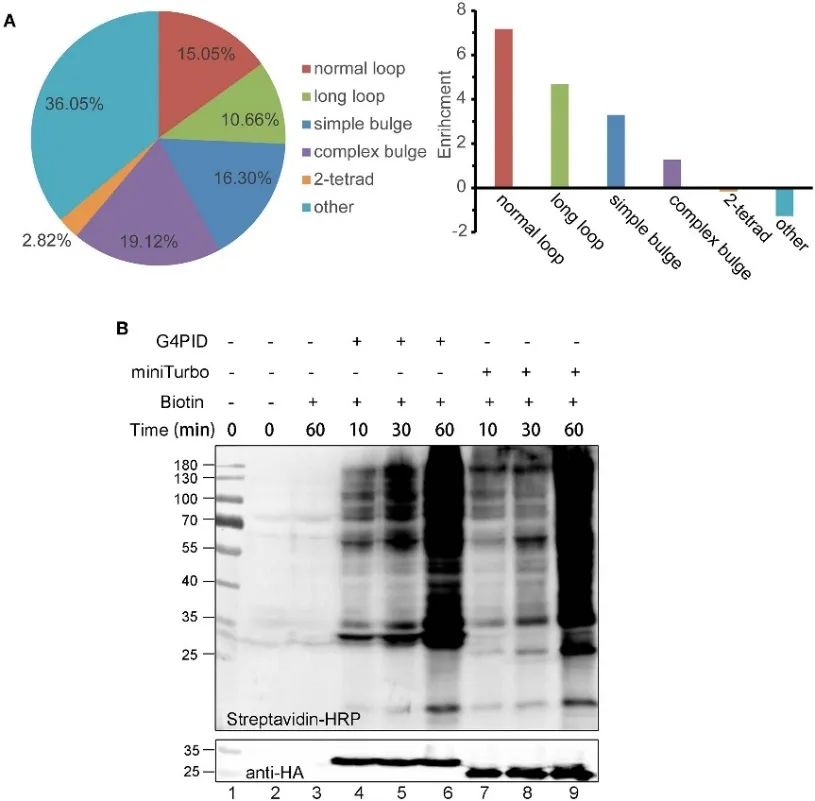
Figuur 2 G 4 PID gecombineerd met RNA G4-experimenten en optimalisatie voor de labelingsefficiëntie in HEK293T-cellen.
3. Het vastleggen en detecteren van het intracellulaire G4-interacterende proteoom met behulp van de PLGPB-methode.
Vervolgens gebruikten de auteurs de G4PID-sonde om het G4-interagerende proteoom in levende cellen te vangen en te detecteren met behulp van de Proximity Labeling of G4-Interacting Proteins (PLGPB) -benadering. In drie onafhankelijke replicaatexperimenten die 578 eiwitten omvatten en een identiteit van 80% bereikten, werd transfectie met G4PID en miniTurbo in HEK293T-cellen uitgevoerd. Screening bracht 149 kandidaat-eiwitten aan het licht die een interactie aangingen met G4, en vertoonden een significante overlap met bestaande databases van G4-interagerende eiwitten. Bovendien duidde de Gene Ontology (GO)-verrijkingsanalyse op een substantiële associatie van deze eiwitten met transcriptionele regulatie, mRNA-splitsing en hermodellering van chromatine, consistent met eerdere bevindingen in G4-onderzoek.
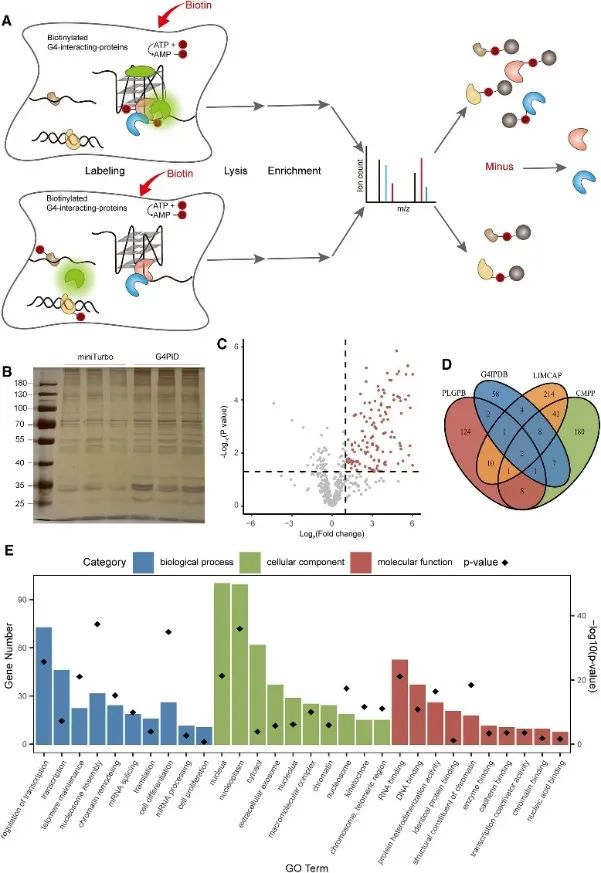
Figuur 3 identificeerde de eiwitten die een interactie aangaan met G4 volgens de PLGPB-methode.
4. Validatie van G4-bindingsaffiniteit bij nieuw geïdentificeerde kandidaten.
Via de PLGPB-methode ontdekten de auteurs een reeks kandidaten die verschillende functionele categorieën omvatten, waaronder gewone telomeer-geassocieerde eiwitten, transcriptiefactoren en relatief ongebruikelijke eiwitbindende eiwitten. Dit geeft aan dat de PLGPB-methode niet alleen eerder erkende G4-interagerende eiwitten opnieuw bevestigt, maar ook het eiwitlandschap aanzienlijk verbreedt door nieuwe kandidaten bloot te leggen die verschillende functionele klassen omspannen. Bovendien valideerden de auteurs het bindingsvermogen van deze kandidaten aan RNA G4 en DNA G4, waarbij een uitgesproken voorkeur voor RNA G4 en variaties in affiniteit voor DNA G4 in verschillende conformaties aan het licht kwamen. Deze bevindingen onderstrepen het potentieel om de eiwit-G4-interactie te moduleren door de G4-conformatie te reguleren.
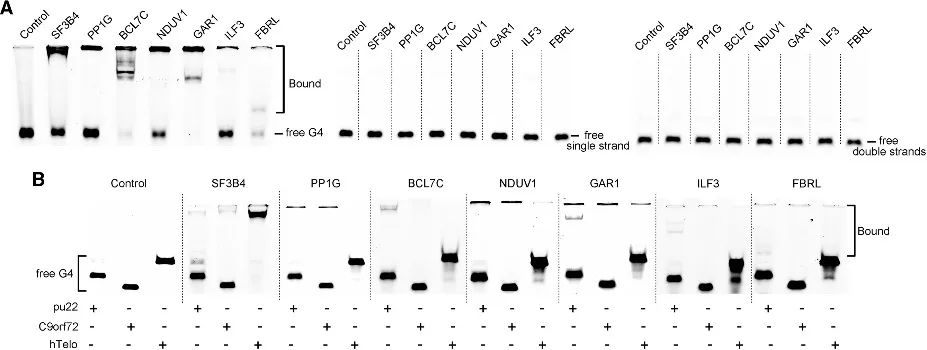
Figuur 4 Interactie tussen de kandidaat-eiwitten en RNA G4 en DNA G4.
5. Bepalen van bindingsplaatsen met hoge betrouwbaarheid van de SF3B4- en G4-structuur.
Tijdens het onderzoek naar de in vivo wijze van G4-binding hebben de auteurs de belangrijke rol van het SF3B4-eiwit blootgelegd. Van SF3B4, bekend om zijn betrokkenheid bij RNA-splitsing, werden de zeer betrouwbare bindingsplaatsen geïdentificeerd door middel van eCLIP-experimenten en een uitgebreide analyse van rG4-seq- en DMS-seq-gegevens. Ongeveer de helft van deze bindingsplaatsen bevatte PQS, voornamelijk geassocieerd met onconventionele G4-structuren. Bovendien observeerden de auteurs een nauwe ruimtelijke relatie tussen SF3B4-bindingsplaatsen en RNA-PQS, met een verrijkte verdeling voornamelijk binnen intronische regio's, vooral proximaal van de 3'-splitsingsplaats. Deze bevindingen suggereren sterk dat de interactie tussen SF3B4 en de G4-structuur een cruciale rol zou kunnen spelen in alternatieve splitsingsmechanismen.
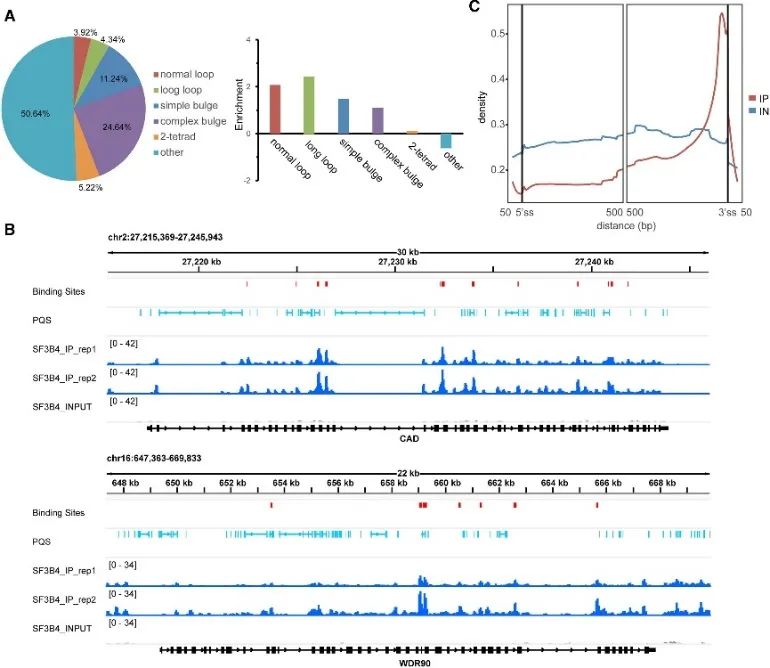
Figuur 5 identificeert de G4-bindingsvoorkeur van SF3B4 in cellen die gebruik maken van eCLIP-technologie.
6. G4-structuur reguleert door SF3B4 gemedieerde alternatieve splitsing.
De auteurs voerden een analyse uit van SF3B4-bindingsplaatsen om de associatie ervan met de G4-structuur te onthullen en onderzochten verder het regulerende mechanisme ervan bij alternatieve splitsingsgebeurtenissen. Conventionele PQS betrokken bij alternatieve splitsing werden gescreend, waarbij twee genen, INPPL1 en PPP6R2, werden geselecteerd voor gedetailleerd onderzoek. G4-structuren binnen INPPL1 en PPP6R2, G4-INPPL1 en G4-PPP6R2 genoemd, werden bevestigd en er werd geïdentificeerd dat SF3B4 specifiek aan deze G4-structuren bindt. Bovendien werd aangetoond dat de toevoeging van het G4-ligand pyridostatine (PDS) de binding van SF3B4 aan G4-structuren vermindert, waardoor exon-inclusiegebeurtenissen tijdens INPPL1- en PPP6R2-mRNA-splitsing worden bevorderd. Deze bevindingen duiden op het potentiële belang van de G4-structuur bij SF3B4-gemedieerde alternatieve splitsing.
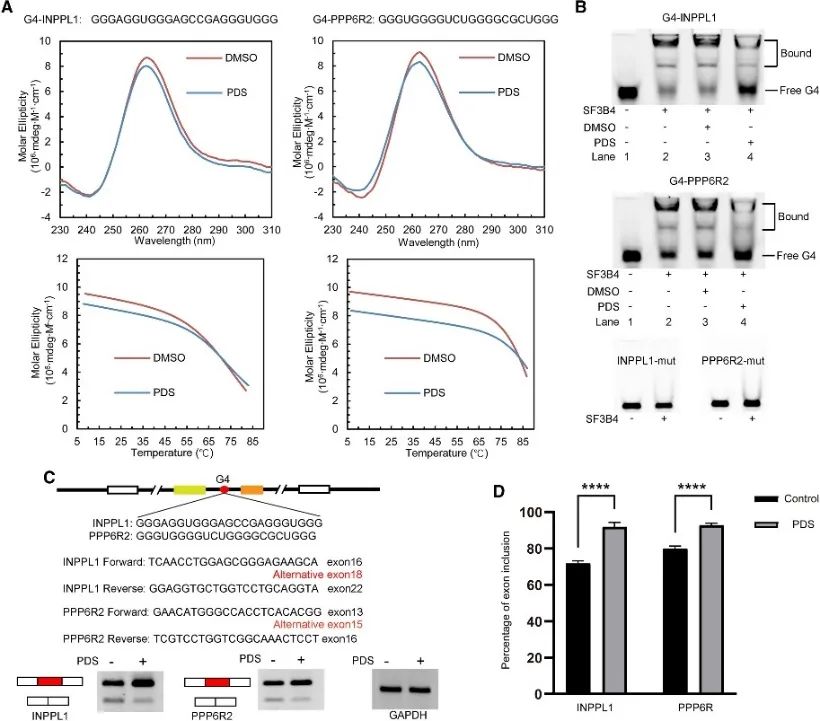
Figuur 6 SF3B4 bindt de G4-structuur en beïnvloedt de alternatieve splitsing van het mRNA.
Concluderend introduceert deze studie een efficiënte toolkit bestaande uit de G4PID-sonde en de PLGPB-strategie, die licht werpt op hoe de wisselwerking tussen G4 en eiwitten in de cel de genexpressie beïnvloedt, en met name het cruciale proces van alternatieve splitsing beïnvloedt. Deze bevindingen verdiepen ons begrip van de regulerende mechanismen waarbij G4 betrokken is bij cellulaire biologische processen en maken de weg vrij voor verder onderzoek naar therapeutische strategieën voor G4-gerelateerde ziekten.

