Interakcje kwasu nukleinowego i białka ujawnione dzięki strategii znakowania zbliżeniowego: badanie sond G4PID i strategii PLGPB
W artykule przedstawiono innowacyjne badanie, w którym autorzy wprowadzają nową dwufunkcyjną sondę o nazwie G4PID. Sonda ta łączy domenę wiążącą G4 (RHAU23) RHAU z ligazą biotynową miniTurbo, umożliwiając precyzyjne namierzanie kwadrupleksów G (G4) i znakowanie białek oddziałujących z G4 w żywych komórkach. Struktury G4, utworzone przez sekwencje kwasów nukleinowych bogatych w guaninę, odgrywają kluczową rolę w różnych procesach komórkowych, w tym w regulacji genów, replikacji DNA i naprawie. Pomimo powszechnego uznania znaczenia G4, specyficzne mechanizmy w komórkach i ich interakcje z białkami pozostają w dużej mierze niezbadane. Wykorzystując G4PID, autorzy opracowali specjalistyczną metodę znakowania biotyną (PLGPB) w celu dokładnej identyfikacji i analizy białek oddziałujących z G4, rzucając światło na kluczową rolę struktur G4 w funkcjach komórkowych. W ramach tego podejścia z powodzeniem zidentyfikowano 149 kandydatów na białka oddziałujące z G4, zaangażowanych głównie w regulację transkrypcji, składanie mRNA i przebudowę chromatyny. Walidacja siedmiu kandydatów na białka ujawniła ich preferencje dotyczące wiązania RNA G4, aczkolwiek z różnym powinowactwem do DNA G4. Szczegółowe badanie czynnika splicingu SF3B4 wykazało jego interakcję ze strukturami G4, wpływając na zdarzenia alternatywnego splicingu. Stabilizacja struktur G4 zmieniła skuteczność wiązania SF3B4 i wpłynęła na alternatywny splicing specyficznych genów (np. INPPL1 i PPP6R2).

Dużą uwagę przykuwa berberyna, tradycyjny alkaloid o szerokim działaniu farmakologicznym, w tym przeciwzapalnym, hipoglikemicznym i chroniącym układ sercowo-naczyniowy. Jednak jego dokładne mechanizmy molekularne, szczególnie w tłumieniu stanu zapalnego, pozostają niejasne.
Niniejsze badanie wypełnia tę lukę w wiedzy, wykorzystując technologię ABPP do zidentyfikowania EIF2AK2 jako krytycznego celu zaangażowanego przez berberynę w jej działaniu przeciwzapalnym. Odkrycia pogłębiają naszą wiedzę na temat mechanizmu działania berberyny i dostarczają naukowych podstaw do zmiany położenia berberyny i opracowania nowych leków przeciwzapalnych ukierunkowanych na EIF2AK2.
Zespół zastosował zaawansowane metody chemoproteomiczne do systematycznego badania interakcji berberyny z białkami wewnątrzkomórkowymi, potwierdzając jej specyficzne wiązanie z EIF2AK2 i modulację jej aktywności enzymatycznej. Wpływa to na szlaki odpowiedzi zapalnej, skutecznie hamując postęp stanu zapalnego. Ten znaczący przełom zapewnia wgląd w mechanizm przeciwzapalny berberyny i wspiera rozwój nowych terapii ukierunkowanych na EIF2AK2.
ChomiX świadczy najnowocześniejsze usługi, takie jak ABPP i CETSA, aby pomóc badaczom w badaniu mechanizmów działania leków i przyspieszaniu prac nad nowymi lekami.
Trasa Badawcza

Proces eksperymentalny
1. Weryfikacja budowy i specyficzności sondy G4PID.
Autorzy początkowo opracowali sondę G4PID z domeną wiążącą G4 RHAU23 umieszczoną na N-końcu i miniTurbo na C-końcu, połączoną elastycznym łącznikiem. Ekspresję i oczyszczanie przeprowadzono przy użyciu szczepu BL21 (DE3). Eksperymenty sprawdzające specyficzne wiązanie z oczyszczonym G4PID ujawniły jego dokładne wiązanie z BCL2 G4, wykazując wartość Kd wynoszącą 15 ± 7 nM, wykazując jednocześnie znikome powinowactwo do innych struktur innych niż G4. Ponadto autorzy ocenili specyficzność biotynylacji G4PID, wykazując jego zdolność do selektywnego biotynylowania białek oddziałujących z G4.
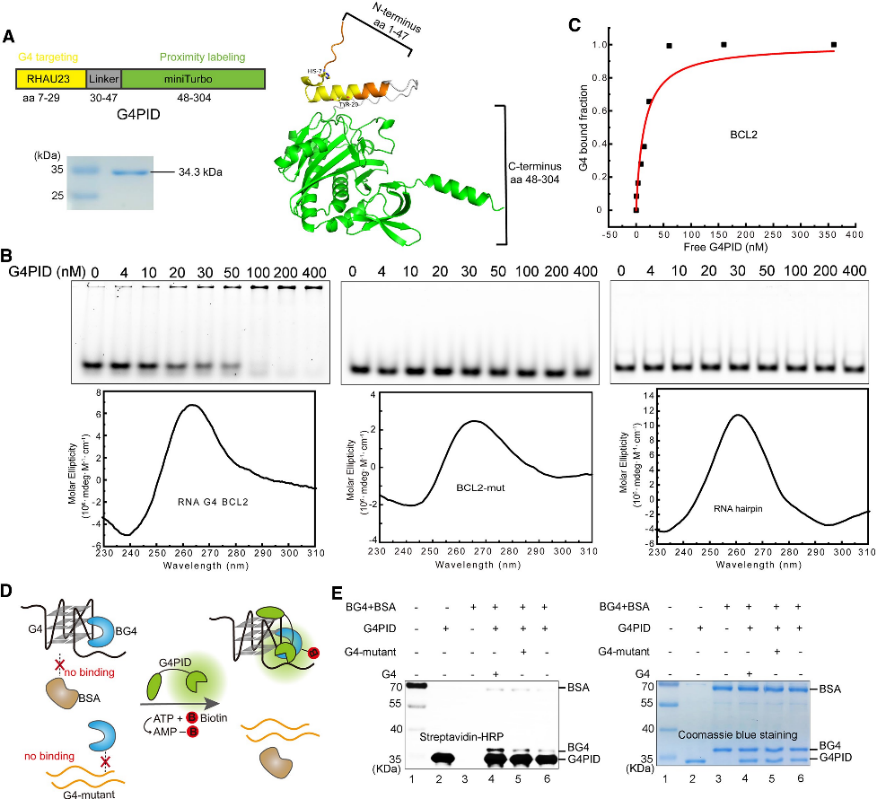
Rycina 1 G4 PID pośredniczy w specyficznej biotynylacji białka oddziałującego z G4
2. Zdolność wiązania RNA G4 przez G4PID w komórkach i charakterystyka znakowania biotyną.
Autorzy stworzyli linie komórkowe HA-G4PID i HA-miniTurbo HeLa w celu zbadania powinowactwa wiązania G4PID z wewnątrzkomórkowym RNA G4 za pomocą eksperymentów eCLIP. Wyniki pokazują selektywne oddziaływanie G4PID z RNA G4, wykazując znacznie wyższe powinowactwo wiązania z sekwencjami RNA zawierającymi PQS w porównaniu z sekwencjami losowymi. Dodatkowo autorzy zaobserwowali unikalne cechy G4PID ułatwiające inicjację znakowania biotyną w komórkach bez konieczności stosowania kofaktorów. Doprowadziło to do znacznej poprawy efektywności znakowania białek, z wyraźnymi sygnałami biotynylacji wykrytymi dla G4PID w ciągu 30 minut, co wskazuje na szybszą kinetykę znakowania w porównaniu z miniTurbo.
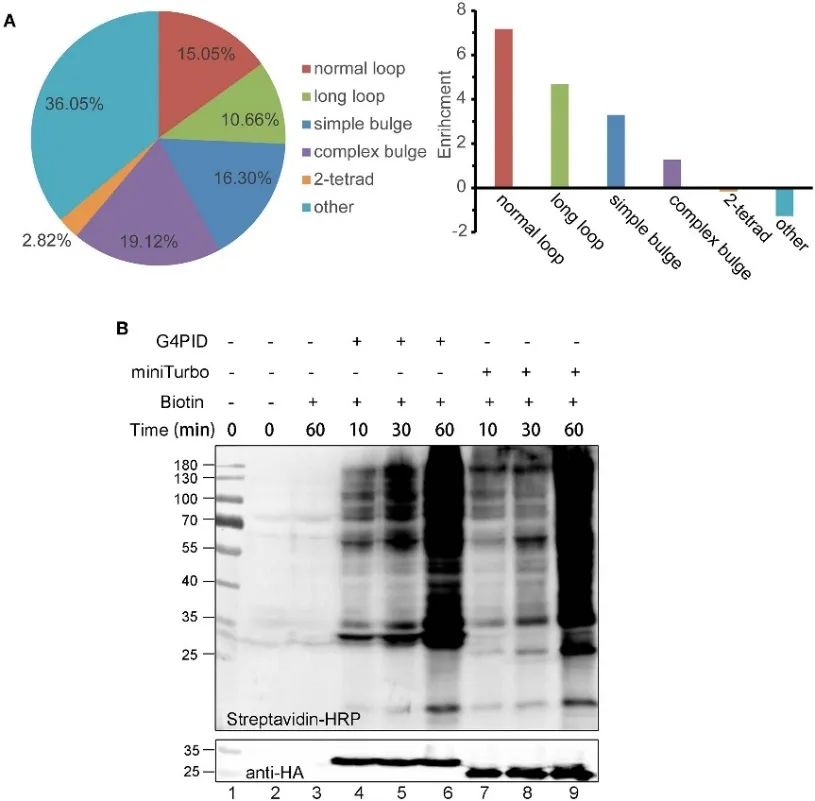
Rysunek 2 G4 PID w połączeniu z eksperymentami RNA G4 i optymalizacją pod kątem wydajności znakowania w komórkach HEK293T.
3. Wychwytywanie i wykrywanie wewnątrzkomórkowego proteomu oddziałującego z G4 przy użyciu metody PLGPB.
Następnie autorzy wykorzystali sondę G4PID do wychwytywania i wykrywania proteomu oddziałującego z G4 w żywych komórkach przy użyciu podejścia Proximity Labelling of G4-Interacting Proteins (PLGPB). W trzech niezależnych powtórzonych eksperymentach obejmujących 578 białek, uzyskując 80% identyczności, przeprowadzono transfekcję G4PID i miniTurbo w komórkach HEK293T. Badanie przesiewowe ujawniło 149 kandydatów na białka oddziałujące z G4, co w znacznym stopniu pokrywa się z istniejącymi bazami danych białek oddziałujących z G4. Co więcej, analiza wzbogacenia Gene Ontology (GO) wykazała istotny związek tych białek z regulacją transkrypcji, składaniem mRNA i przebudową chromatyny, zgodnie z wcześniejszymi ustaleniami z badania G4.
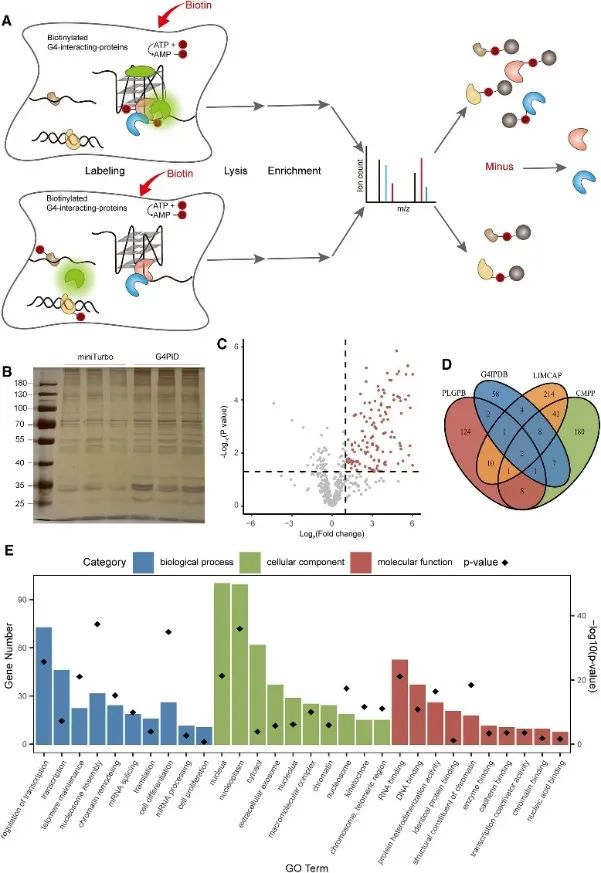
Na Figurze 3 zidentyfikowano białka oddziałujące z G4 metodą PLGPB.
4. Walidacja powinowactwa wiązania G4 u nowo zidentyfikowanych kandydatów.
Dzięki metodzie PLGPB autorzy odkryli szereg kandydatów obejmujących różne kategorie funkcjonalne, w tym popularne białka związane z telomerami, czynniki transkrypcyjne i stosunkowo rzadkie białka wiążące białka. Wskazuje to, że metoda PLGPB nie tylko potwierdza wcześniej uznane białka oddziałujące z G4, ale także znacząco poszerza krajobraz białek poprzez odkrywanie nowych kandydatów obejmujących różne klasy funkcjonalne. Ponadto autorzy sprawdzili zdolność wiązania tych kandydatów z RNA G4 i DNA G4, ujawniając wyraźną preferencję dla RNA G4 i różnice w powinowactwie do DNA G4 w różnych konformacjach. Odkrycia te podkreślają potencjał modulowania interakcji białko-G4 poprzez regulację konformacji G4.
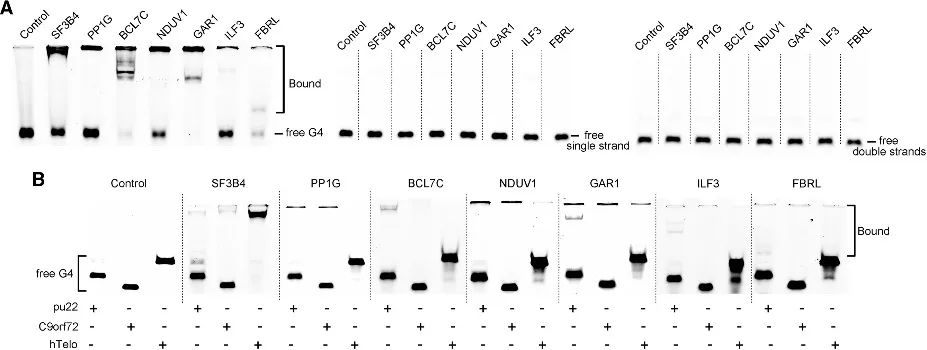
Rycina 4 Interakcja między białkami kandydującymi a RNA G4 i DNA G4.
5. Wyznaczanie miejsc wiążących o wysokim stopniu ufności w strukturze SF3B4 i G4.
Podczas badania sposobu wiązania G4 in vivo autorzy odkryli znaczącą rolę białka SF3B4. Miejsca wiązania SF3B4, znanego ze swojego zaangażowania w składanie RNA, zidentyfikowano w eksperymentach eCLIP i kompleksowej analizie danych rG4-seq i DMS-seq. Około połowa tych miejsc wiązania zawierała PQS, głównie związaną z niekonwencjonalnymi strukturami G4. Co więcej, autorzy zaobserwowali ścisły związek przestrzenny między miejscami wiązania SF3B4 i PQS RNA, ze wzbogaconą dystrybucją głównie w regionach intronowych, szczególnie w pobliżu miejsca splicingu 3'. Odkrycia te zdecydowanie sugerują, że interakcja między SF3B4 i strukturą G4 może odgrywać kluczową rolę w alternatywnych mechanizmach splicingu.
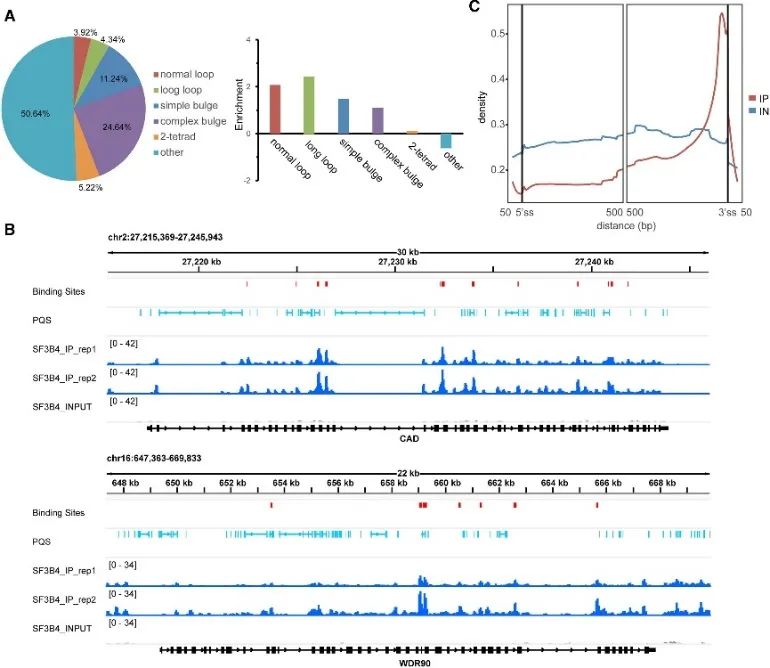
Figura 5 przedstawia preferencję wiązania G4 SF3B4 w komórkach wykorzystujących technologię eCLIP.
6. Struktura G4 reguluje alternatywny splicing za pośrednictwem SF3B4.
Autorzy przeprowadzili analizę miejsc wiązania SF3B4, aby odkryć jego powiązanie ze strukturą G4 i dokładniej zbadali jego mechanizm regulacyjny w zdarzeniach alternatywnego splicingu. Przeszukano konwencjonalne PQS związane z alternatywnym splicingiem, wybierając do szczegółowego badania dwa geny, INPPL1 i PPP6R2. Potwierdzono struktury G4 w obrębie INPPL1 i PPP6R2, zwane G4-INPPL1 i G4-PPP6R2, a SF3B4 zidentyfikowano jako specyficznie wiążący te struktury G4. Ponadto wykazano, że dodatek pirydostatyny ligandu G4 (PDS) zmniejsza wiązanie SF3B4 ze strukturami G4, w konsekwencji promując zdarzenia inkluzji egzonów podczas splicingu mRNA INPPL1 i PPP6R2. Odkrycia te wskazują na potencjalne znaczenie struktury G4 w alternatywnym splicingu za pośrednictwem SF3B4.
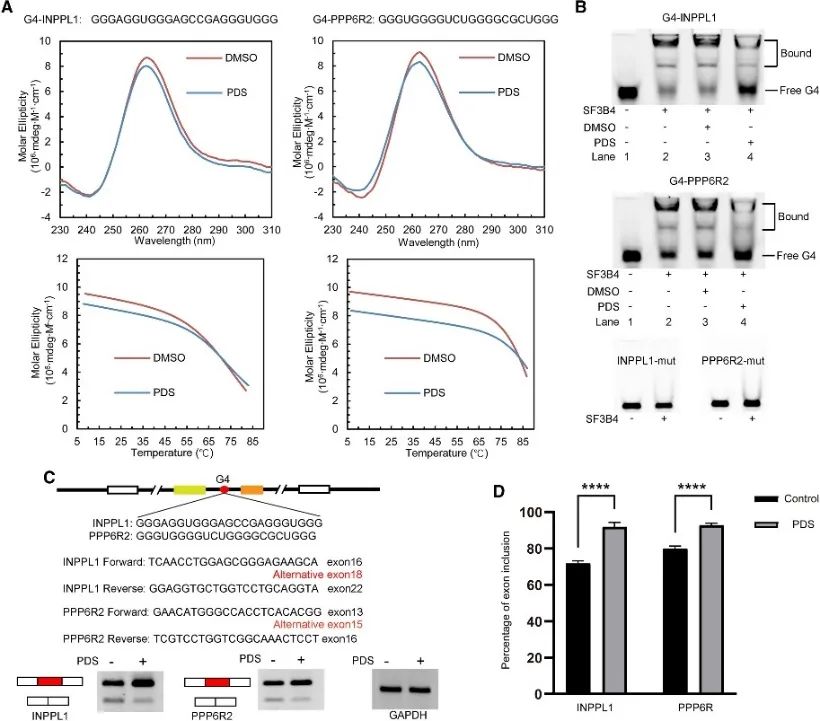
Figura 6 SF3B4 wiąże strukturę G4 i wpływa na alternatywny splicing mRNA.
Podsumowując, badanie to wprowadza skuteczny zestaw narzędzi obejmujący sondę G4PID i strategię PLGPB, rzucając światło na to, jak wzajemne oddziaływanie pomiędzy G4 i białkami w komórce wpływa na ekspresję genów, szczególnie wpływając na kluczowy proces alternatywnego splicingu. Odkrycia te pogłębiają nasze zrozumienie mechanizmów regulacyjnych obejmujących G4 w komórkowych procesach biologicznych i torują drogę do dalszych badań nad strategiami terapeutycznymi chorób związanych z G4.

