Interações ácido nucleico-proteína reveladas por meio da estratégia de rotulagem de proximidade: explorando sondas G4PID e estratégia PLGPB
Este artigo apresenta um estudo inovador em que os autores apresentam uma nova sonda bifuncional denominada G4PID. Esta sonda combina o domínio de ligação G4 (RHAU23) de RHAU com a miniTurbo biotina ligase, permitindo o direcionamento preciso de G-quadruplexes (G4) e marcação de proteínas que interagem com G4 em células vivas. As estruturas G4, formadas por sequências de ácidos nucleicos ricas em guanina, são essenciais em vários processos celulares, incluindo regulação genética, replicação e reparação de DNA. Apesar do amplo reconhecimento da importância do G4, os mecanismos específicos dentro das células e as suas interações com as proteínas permanecem largamente inexplorados. Empregando o G4PID, os autores desenvolveram um método especializado de marcação de biotina (PLGPB) para identificar e analisar com precisão as proteínas que interagem com o G4, esclarecendo o papel crucial das estruturas G4 nas funções celulares. Esta abordagem identificou com sucesso 149 proteínas candidatas interagindo com G4, predominantemente envolvidas na regulação transcricional, splicing de mRNA e remodelação da cromatina. A validação de sete proteínas candidatas revelou a sua preferência pela ligação ao ARN G4, embora com afinidades variadas para o ADN G4. O exame detalhado do fator de splicing SF3B4 demonstrou sua interação com estruturas G4, impactando eventos de splicing alternativos. A estabilização das estruturas G4 alterou a eficiência de ligação do SF3B4 e influenciou o splicing alternativo de genes específicos (por exemplo, INPPL1 e PPP6R2).

A berberina, um alcalóide tradicional com amplos efeitos farmacológicos, incluindo antiinflamatório, hipoglicemia e proteção cardiovascular, tem atraído atenção considerável. No entanto, os seus mecanismos moleculares precisos, particularmente na supressão da inflamação, permanecem obscuros.
Este estudo preenche esta lacuna de conhecimento utilizando a tecnologia ABPP para identificar o EIF2AK2 como um alvo crítico envolvido pela berberina pela sua ação anti-inflamatória. As descobertas aprofundam a nossa compreensão do mecanismo da berberina e fornecem uma base científica para o reposicionamento da berberina e o desenvolvimento de novos medicamentos anti-inflamatórios direcionados ao EIF2AK2.
A equipa empregou métodos quimioproteómicos avançados para investigar sistematicamente as interacções da berberina com proteínas intracelulares, confirmando a sua ligação específica ao EIF2AK2 e a modulação da sua actividade enzimática. Isto influencia as vias de resposta inflamatória, inibindo eficazmente a progressão da inflamação. Este avanço significativo oferece informações sobre o mecanismo anti-inflamatório da berberina e apoia o desenvolvimento de novas terapias direcionadas ao EIF2AK2.
A ChomiX fornece serviços de ponta como ABPP e CETSA para ajudar os pesquisadores na exploração de mecanismos de medicamentos e na aceleração dos esforços de desenvolvimento de novos medicamentos.
Rota de Pesquisa

Processo experimental
1. Verificação de Construção e Especificidade da Sonda G4PID.
Os autores projetaram inicialmente a sonda G4PID, com o domínio de ligação G4 RHAU23 posicionado no terminal N e o miniTurbo no terminal C, ligado por um ligante flexível. A expressão e purificação foram realizadas utilizando a cepa BL21 (DE3). Experimentos de validação de ligação específica com G4PID purificado revelaram sua ligação precisa ao BCL2 G4, exibindo um valor Kd de 15 ± 7 nM, enquanto exibia afinidade insignificante para outras estruturas não-G4. Além disso, os autores avaliaram a especificidade da biotinilação do G4PID, demonstrando a sua capacidade de biotinilar seletivamente proteínas que interagem com o G4.
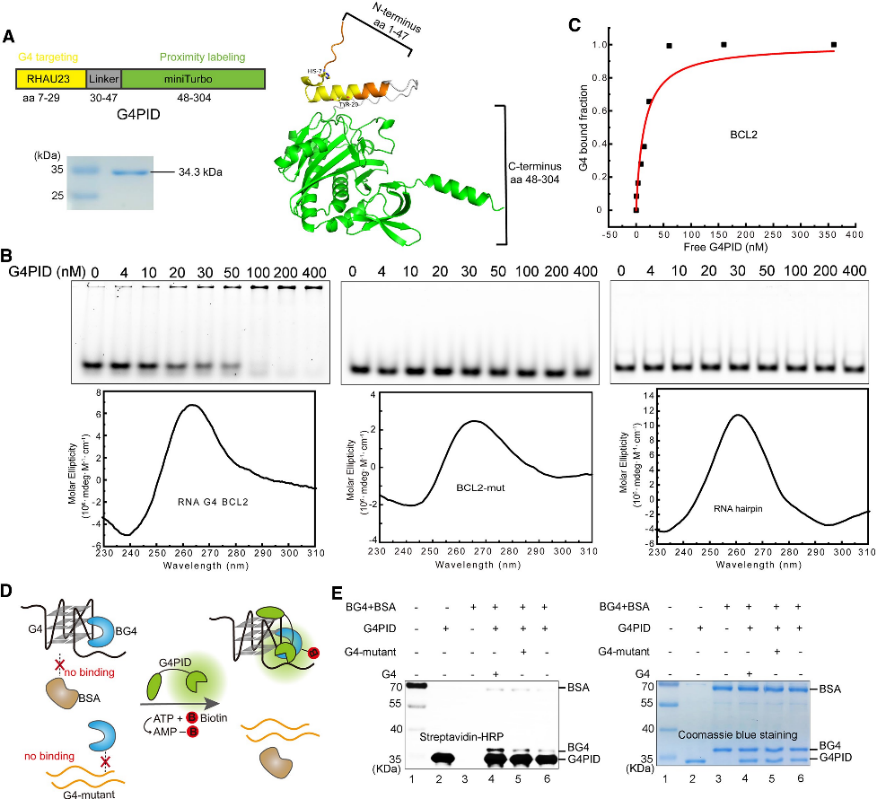
Figura 1 G4 PID medeia a biotinilação específica da proteína que interage com G 4
2. Capacidade de ligação ao RNA G4 de G4PID em células e características de marcação de biotina.
Os autores estabeleceram linhas celulares HA-G4PID e HA-miniTurbo HeLa para investigar a afinidade de ligação do G4PID ao RNA intracelular G4 através de experimentos eCLIP. Os resultados demonstram a interação seletiva de G4PID com RNA G4, exibindo afinidade de ligação notavelmente maior a sequências de RNA contendo PQS em comparação com sequências aleatórias. Além disso, os autores observaram características únicas do G4PID facilitando o início da marcação da biotina nas células sem a necessidade de cofatores. Isto levou a um aumento significativo na eficiência da marcação de proteínas, com sinais evidentes de biotinilação detectados para G4PID em 30 minutos, indicando uma cinética de marcação mais rápida em comparação com o miniTurbo.
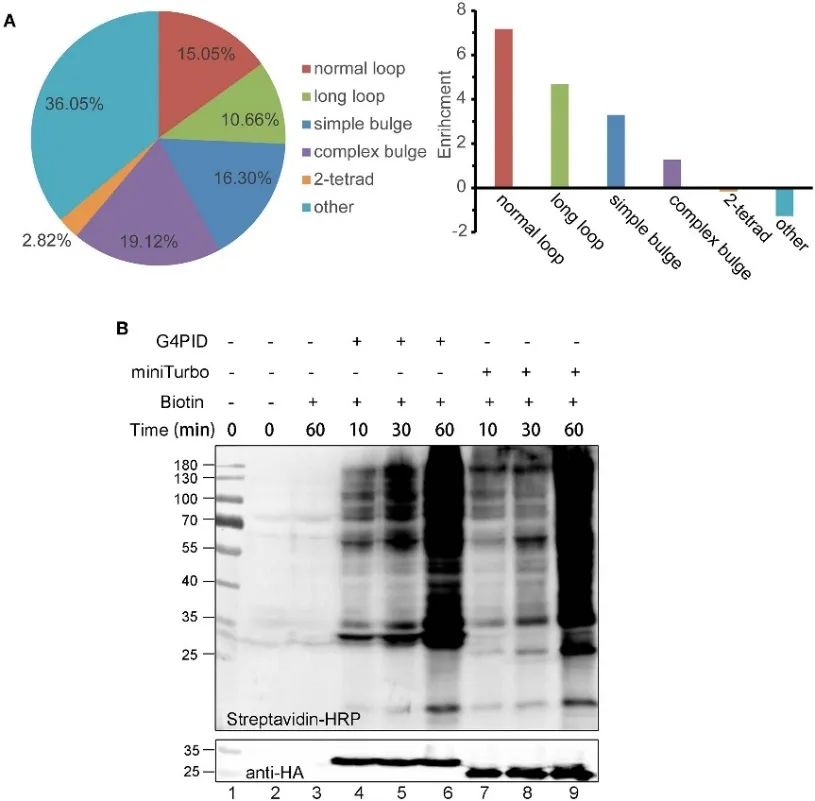
Figura 2 G 4 PID combinado com experimentos de RNA G4 e otimização para eficiência de marcação em células HEK293T.
3. Capturando e detectando o proteoma intracelular G4 interagindo usando o método PLGPB.
Posteriormente, os autores empregaram a sonda G4PID para capturar e detectar o proteoma que interage com G4 dentro de células vivas usando a abordagem de marcação de proximidade de proteínas que interagem com G4 (PLGPB). Em três experiências replicadas independentes abrangendo 578 proteínas, alcançando uma identidade de 80%, foi realizada transfecção com G4PID e miniTurbo em células HEK293T. A triagem revelou 149 proteínas candidatas interagindo com G4, exibindo sobreposição significativa com bancos de dados existentes de proteínas que interagem com G4. Além disso, a análise de enriquecimento da Gene Ontology (GO) indicou uma associação substancial destas proteínas com regulação transcricional, splicing de mRNA e remodelação da cromatina, consistente com descobertas anteriores na pesquisa do G4.
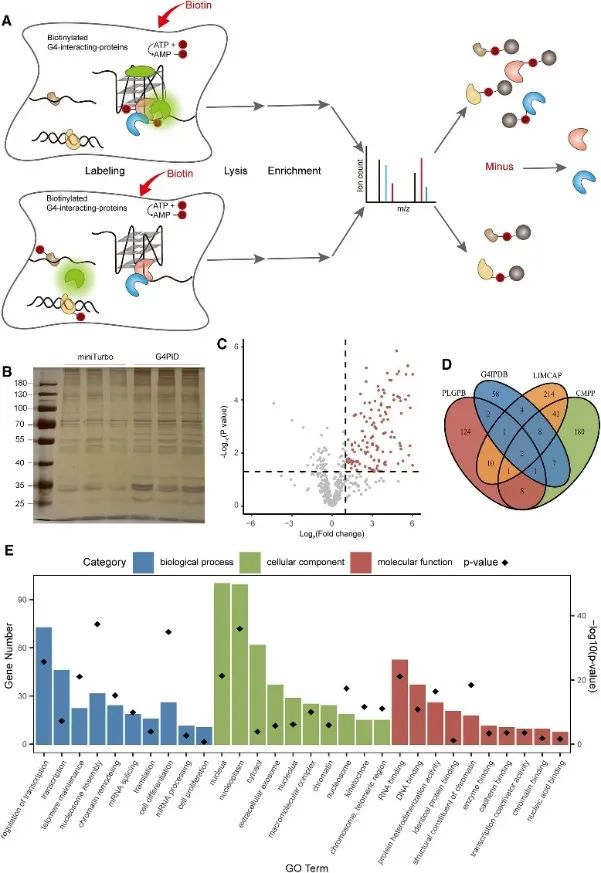
A Figura 3 identificou as proteínas interagindo com G4 pelo método PLGPB.
4. Validação da Afinidade Vinculante G4 em Candidatos Recentemente Identificados.
Através do método PLGPB, os autores descobriram uma gama de candidatos abrangendo várias categorias funcionais, incluindo proteínas comuns associadas aos telômeros, fatores de transcrição e proteínas de ligação a proteínas relativamente incomuns. Isto indica que o método PLGPB não apenas reafirma proteínas previamente reconhecidas que interagem com G4, mas também amplia significativamente o panorama proteico ao descobrir novos candidatos abrangendo diversas classes funcionais. Além disso, os autores validaram a capacidade de ligação destes candidatos ao RNA G4 e ao DNA G4, revelando uma preferência pronunciada pelo RNA G4 e variações na afinidade pelo DNA G4 em diferentes conformações. Estas descobertas sublinham o potencial para modular a interacção proteína-G4 através da regulação da conformação G4.
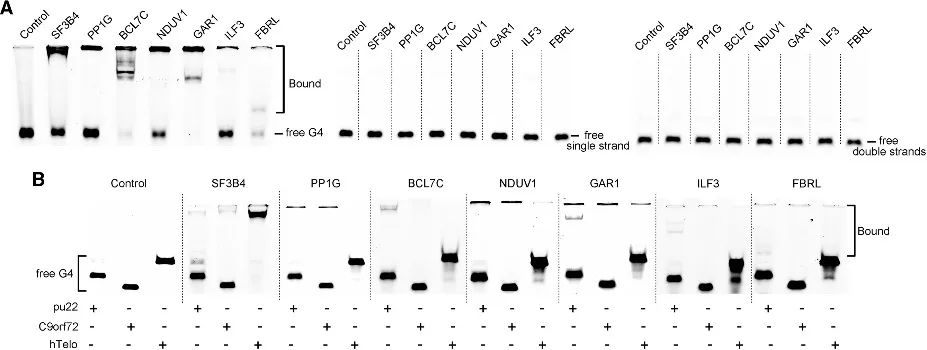
Figura 4 Interação entre as proteínas candidatas e RNA G4 e DNA G4.
5. Determinação de locais de ligação de alta confiança da estrutura SF3B4 e G4.
Durante a investigação sobre o modo in vivo de ligação ao G4, os autores descobriram o papel significativo da proteína SF3B4. SF3B4, conhecido por seu envolvimento no splicing de RNA, teve seus locais de ligação de alta confiança identificados por meio de experimentos eCLIP e uma análise abrangente de dados de rG4-seq e DMS-seq. Aproximadamente metade destes locais de ligação continham PQS, predominantemente associados a estruturas G4 não convencionais. Além disso, os autores observaram uma estreita relação espacial entre os locais de ligação do SF3B4 e o RNA PQS, com uma distribuição enriquecida principalmente nas regiões intrônicas, particularmente proximais ao local de splice 3'. Estas descobertas sugerem fortemente que a interação entre o SF3B4 e a estrutura G4 pode desempenhar um papel crucial em mecanismos de splicing alternativos.
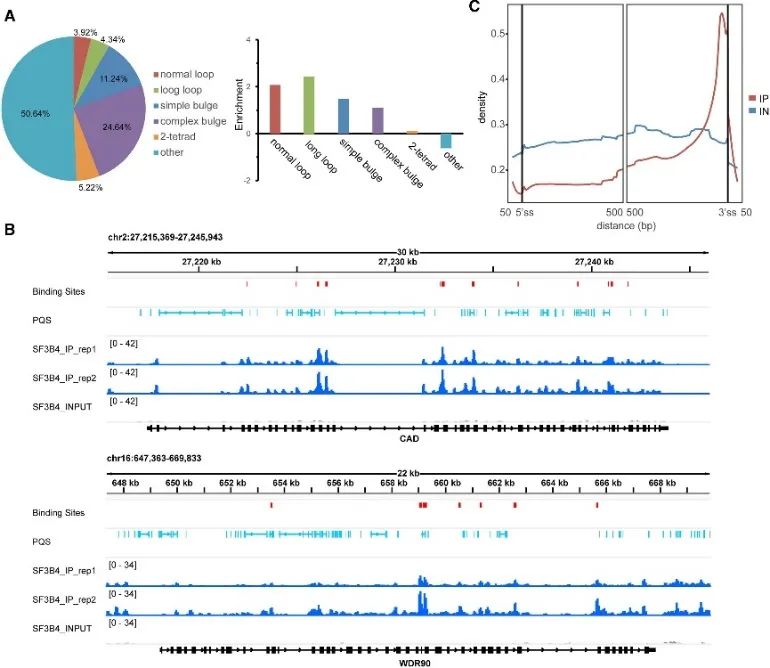
A Figura 5 identifica a preferência de ligação G4 do SF3B4 em células usando a tecnologia eCLIP.
6. A estrutura G4 regula o splicing alternativo mediado por SF3B4.
Os autores conduziram uma análise dos locais de ligação do SF3B4 para revelar a sua associação com a estrutura G4 e investigaram ainda mais o seu mecanismo regulador em eventos de splicing alternativos. Os PQS convencionais implicados no splicing alternativo foram selecionados, com dois genes, INPPL1 e PPP6R2, selecionados para exame detalhado. As estruturas G4 dentro de INPPL1 e PPP6R2, denominadas G4-INPPL1 e G4-PPP6R2, foram confirmadas, e SF3B4 foi identificado como ligando-se especificamente a estas estruturas G4. Além disso, foi demonstrado que a adição do ligante G4 piridostatina (PDS) diminui a ligação do SF3B4 às estruturas G4, promovendo consequentemente eventos de inclusão de exon durante o splicing do mRNA de INPPL1 e PPP6R2. Estas descobertas indicam o significado potencial da estrutura G4 no splicing alternativo mediado por SF3B4.
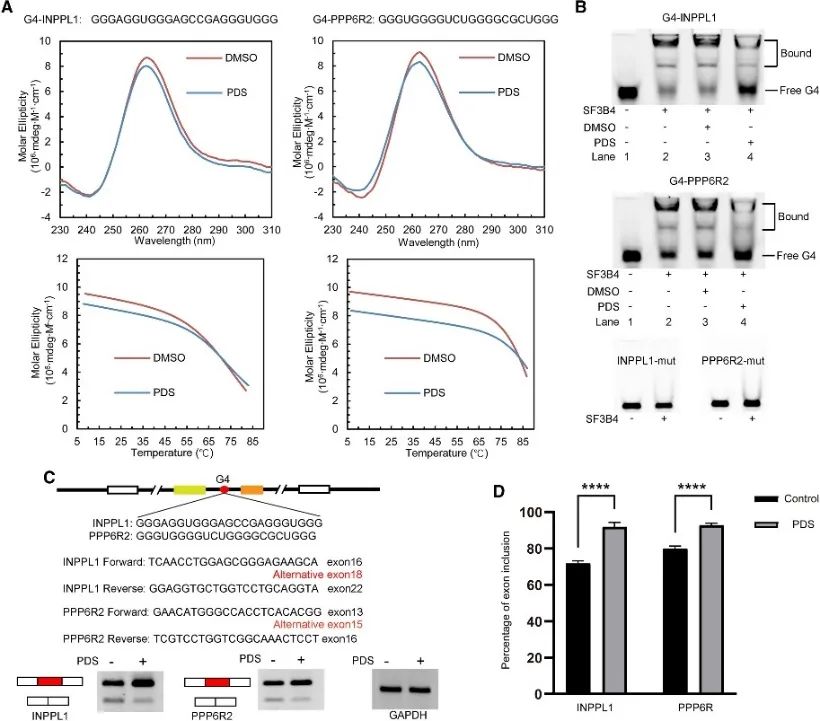
Figura 6 SF3B4 liga-se à estrutura G4 e afeta o splicing alternativo do mRNA.
Em conclusão, este estudo apresenta um kit de ferramentas eficiente que compreende a sonda G4PID e a estratégia PLGPB, esclarecendo como a interação entre G4 e proteínas dentro da célula impacta a expressão gênica, influenciando particularmente o processo crucial de splicing alternativo. Estas descobertas aprofundam a nossa compreensão dos mecanismos reguladores que envolvem o G4 nos processos biológicos celulares e abrem caminho para uma maior exploração de estratégias terapêuticas para doenças relacionadas com o G4.

