Nukleinsyra-proteininteraktioner avslöjade genom strategi för närhetsmärkning: Utforska G4PID-sonder och PLGPB-strategi
Den här artikeln presenterar en innovativ studie där författarna introducerar en ny bifunktionell sond som kallas G4PID. Denna sond kombinerar den G4-bindande domänen (RHAU23) av RHAU med miniTurbo biotinligas, vilket möjliggör exakt inriktning av G-quadruplexes (G4) och taggning av G4-interagerande proteiner i levande celler. G4-strukturer, bildade av guaninrika nukleinsyrasekvenser, är centrala i olika cellulära processer inklusive genreglering, DNA-replikation och reparation. Trots ett utbrett erkännande av G4:s betydelse förblir de specifika mekanismerna inom celler och deras interaktioner med proteiner i stort sett outforskade. Med hjälp av G4PID utvecklade författarna en specialiserad biotinmärkningsmetod (PLGPB) för att exakt identifiera och analysera G4-interagerande proteiner, vilket belyser den avgörande rollen för G4-strukturer i cellulära funktioner. Detta tillvägagångssätt identifierade framgångsrikt 149 proteinkandidater som interagerar med G4, huvudsakligen involverade i transkriptionell reglering, mRNA-splitsning och kromatinremodellering. Validering av sju kandidatproteiner avslöjade deras preferens för RNA G4-bindning, om än med varierande affinitet till DNA G4. Detaljerad undersökning av splitsningsfaktorn SF3B4 visade dess interaktion med G4-strukturer, vilket påverkar alternativa splitsningshändelser. Stabilisering av G4-strukturer förändrade SF3B4:s bindningseffektivitet och påverkade alternativ splitsning av specifika gener (t.ex. INPPL1 och PPP6R2).

Berberin, en traditionell alkaloid med omfattande farmakologiska effekter inklusive antiinflammation, hypoglykemi och kardiovaskulärt skydd, har rönt stor uppmärksamhet. Dess exakta molekylära mekanismer, särskilt vid inflammationsdämpning, förblir dock oklara.
Denna studie fyller denna kunskapslucka med hjälp av ABPP-teknik för att identifiera EIF2AK2 som ett kritiskt mål för berberin för dess antiinflammatoriska verkan. Fynden fördjupar vår förståelse av berberins mekanism och ger en vetenskaplig grund för att ompositionera berberin och utveckla nya EIF2AK2-riktade antiinflammatoriska läkemedel.
Teamet använde avancerade kemoproteomiska metoder för att systematiskt undersöka berberins interaktioner med intracellulära proteiner, vilket bekräftade dess specifika bindning till EIF2AK2 och modulering av dess enzymatiska aktivitet. Detta påverkar inflammatoriska reaktionsvägar, vilket effektivt hämmar inflammationsprogression. Detta betydande genombrott ger insikter i berberins antiinflammatoriska mekanism och stödjer utvecklingen av nya behandlingar riktade mot EIF2AK2.
ChomiX tillhandahåller banbrytande tjänster som ABPP och CETSA för att hjälpa forskare att utforska läkemedelsmekanismer och påskynda nya läkemedelsutvecklingsinsatser.
Forskningsväg

Experimentell process
1. Konstruktion och specificitetsverifiering av G4PID-sonden.
Författarna konstruerade initialt G4PID-proben, med den G4-bindande domänen RHAU23 placerad vid N-terminalen och miniTurbo vid C-terminalen, länkad med en flexibel länk. Expression och rening utfördes med användning av BL21 (DE3)-stammen. Specifika bindningsvalideringsexperiment med renat G4PID avslöjade dess exakta bindning till BCL2 G4, uppvisande ett Kd-värde på 15 ± 7 nM, samtidigt som det uppvisade försumbar affinitet mot andra icke-G4-strukturer. Dessutom bedömde författarna biotinyleringsspecificiteten för G4PID, vilket visar dess förmåga att selektivt biotinylera G4-interagerande proteiner.
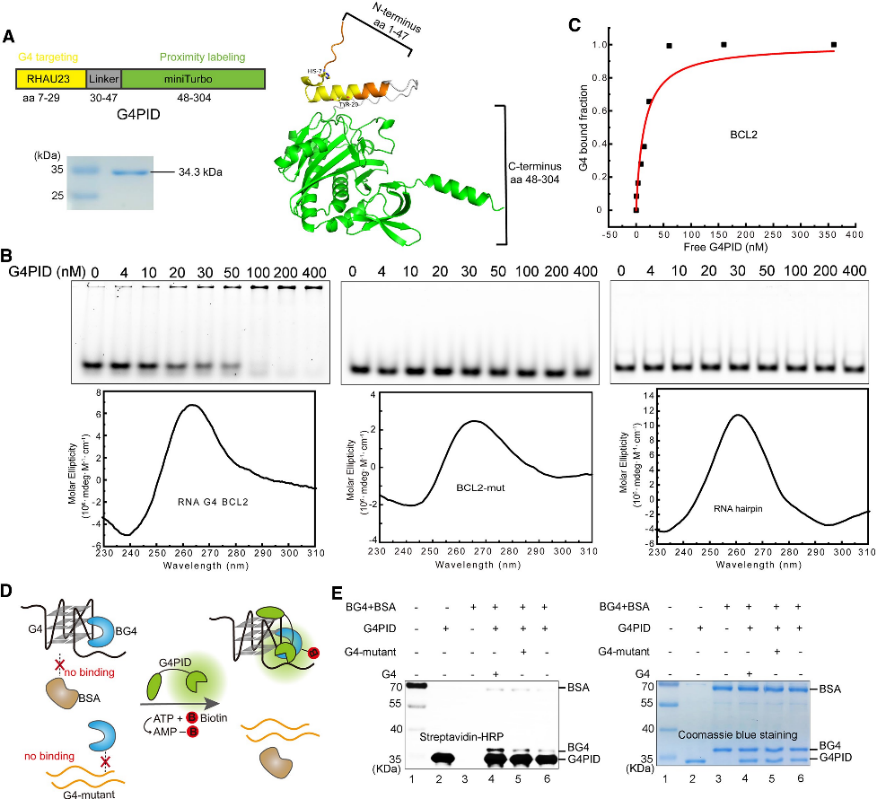
Figur 1 G4 PID förmedlar den specifika biotinyleringen av det G4-interagerande proteinet
2. RNA G4-bindningsförmåga hos G4PID i celler och biotinmärkningsegenskaper.
Författarna etablerade HA-G4PID och HA-miniTurbo HeLa-cellinjer för att undersöka G4PIDs bindningsaffinitet till intracellulärt RNA G4 via eCLIP-experiment. Resultaten visar den selektiva interaktionen av G4PID med RNA G4, som uppvisar avsevärt högre bindningsaffinitet till RNA-sekvenser som innehåller PQS jämfört med slumpmässiga sekvenser. Dessutom observerade författarna unika egenskaper hos G4PID som underlättar initieringen av biotinmärkning i celler utan att kräva kofaktorer. Detta ledde till en betydande förbättring av proteinmärkningseffektiviteten, med tydliga biotinyleringssignaler detekterade för G4PID inom 30 minuter, vilket indikerar snabbare märkningskinetik jämfört med miniTurbo.
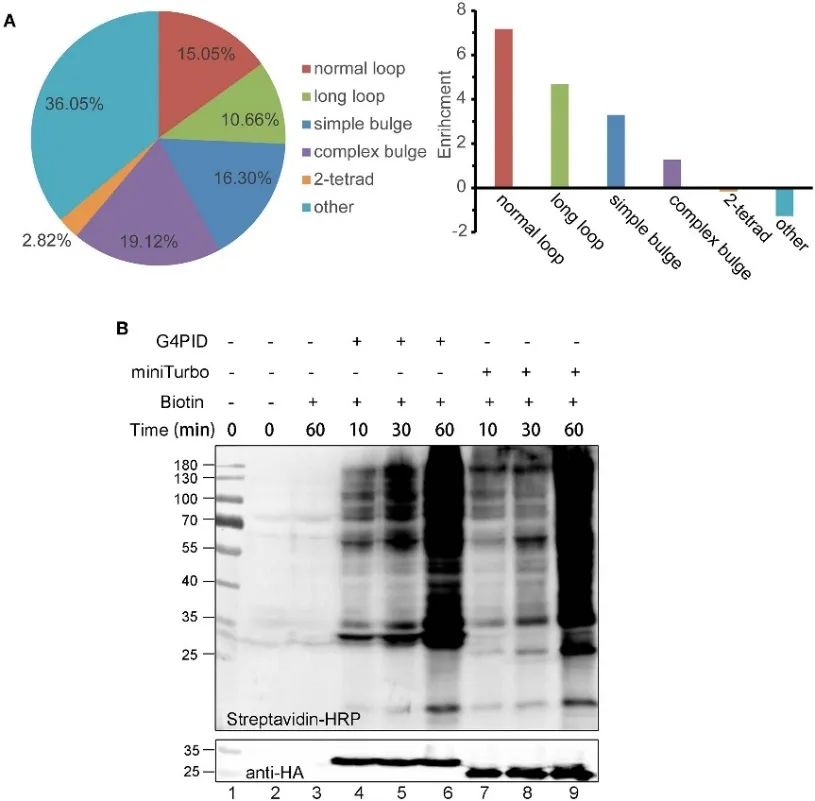
Figur 2 G 4 PID kombinerat med RNA G4-experiment och optimering för märkningseffektiviteten i HEK293T-celler.
3. Fånga och detektera det intracellulära G4-interagerande proteomet med hjälp av PLGPB-metoden.
Därefter använde författarna G4PID-sonden för att fånga och detektera det G4-interagerande proteomet i levande celler med hjälp av Proximity Labeling of G4-Interacting Proteins (PLGPB) tillvägagångssätt. I tre oberoende replikatexperiment som omfattar 578 proteiner, för att uppnå en 80% identitet, utfördes transfektion med G4PID och miniTurbo i HEK293T-celler. Screening avslöjade 149 kandidatproteiner som interagerar med G4, som uppvisar betydande överlappning med befintliga databaser med G4-interagerande proteiner. Dessutom indikerade Gene Ontology (GO) anrikningsanalys en betydande association av dessa proteiner med transkriptionell reglering, mRNA-splitsning och kromatinremodellering, i överensstämmelse med tidigare fynd i G4-forskning.
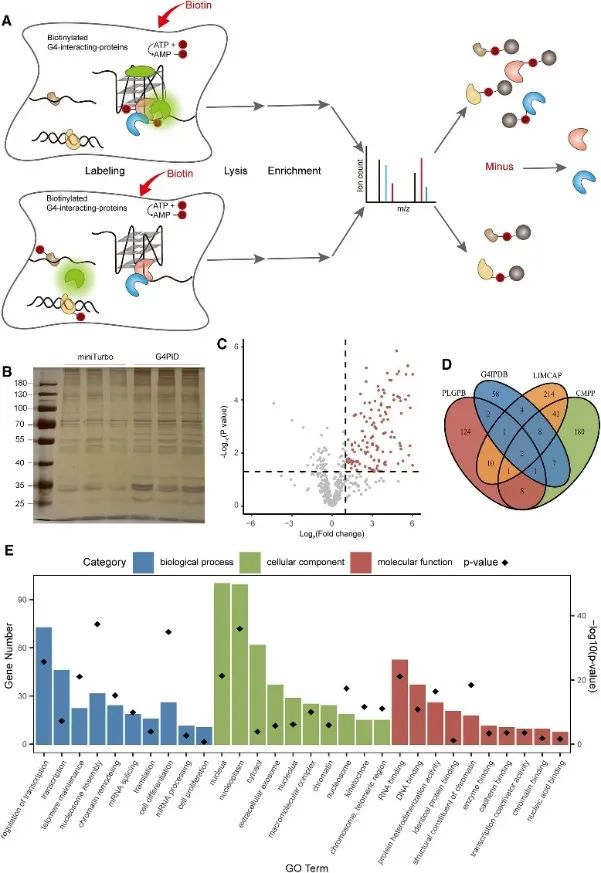
Figur 3 identifierade proteinerna som interagerar med G4 genom PLGPB-metoden.
4. Validering av G4-bindande affinitet hos nyligen identifierade kandidater.
Genom PLGPB-metoden upptäckte författarna en rad kandidater som omfattar olika funktionella kategorier, inklusive vanliga telomerassocierade proteiner, transkriptionsfaktorer och relativt ovanliga proteinbindande proteiner. Detta indikerar att PLGPB-metoden inte bara bekräftar tidigare erkända G4-interagerande proteiner utan också avsevärt breddar proteinlandskapet genom att avslöja nya kandidater som spänner över olika funktionella klasser. Dessutom validerade författarna bindningskapaciteten hos dessa kandidater till RNA G4 och DNA G4, vilket avslöjade en uttalad preferens för RNA G4 och variationer i affinitet till DNA G4 i olika konformationer. Dessa fynd understryker potentialen att modulera protein-G4-interaktion genom att reglera G4-konformation.
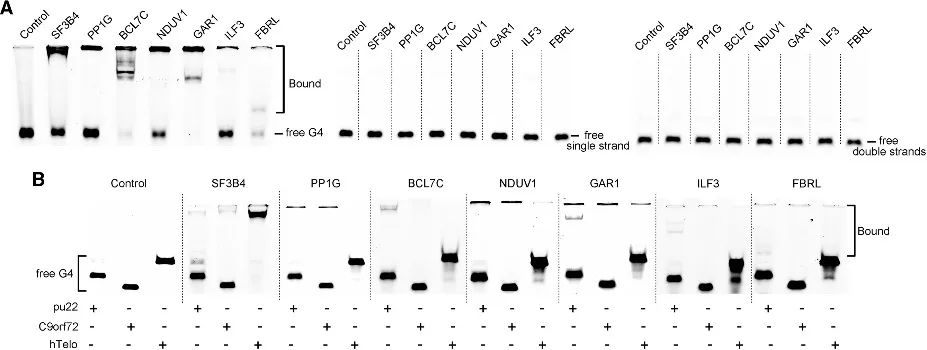
Figur 4 Interaktion mellan kandidatproteinerna och RNA G4 och DNA G4.
5. Fastställa bindningsplatser med hög konfidens för SF3B4- och G4-struktur.
Under undersökningen av in vivo-sättet för G4-bindning, grävde författarna fram den betydande rollen för SF3B4-proteinet. SF3B4, känd för sitt engagemang i RNA-splitsning, fick sina bindningsställen med hög konfidens identifierade genom eCLIP-experiment och en omfattande analys av rG4-seq och DMS-seq-data. Ungefär hälften av dessa bindningsställen innehöll PQS, övervägande associerat med okonventionella G4-strukturer. Dessutom observerade författarna ett nära rumsligt samband mellan SF3B4-bindningsställen och RNA PQS, med en berikad distribution främst inom introniska regioner, särskilt proximalt till 3'-splitsstället. Dessa fynd tyder starkt på att interaktionen mellan SF3B4 och G4-strukturen kan spela en avgörande roll i alternativa splitsningsmekanismer.
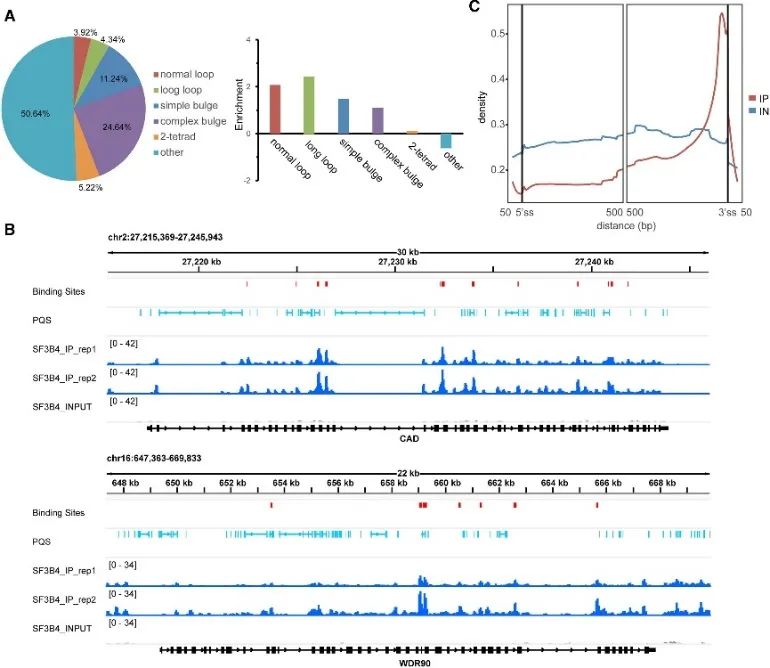
Figur 5 identifierar G4-bindningspreferensen för SF3B4 i celler som använder eCLIP-teknik.
6. G4-struktur reglerar SF3B4-medierad alternativ skarvning.
Författarna genomförde en analys av SF3B4-bindningsställen för att avslöja dess samband med G4-strukturen och undersökte ytterligare dess regleringsmekanism i alternativa splitsningshändelser. Konventionella PQS inblandade i alternativ splitsning screenades, med två gener, INPPL1 och PPP6R2, valda för detaljerad undersökning. G4-strukturer inom INPPL1 och PPP6R2, benämnda G4-INPPL1 och G4-PPP6R2, bekräftades och SF3B4 identifierades som specifikt bindande till dessa G4-strukturer. Dessutom visades det att tillsatsen av G4-liganden pyridostatin (PDS) minskar SF3B4-bindning till G4-strukturer, vilket följaktligen främjar exoninklusionshändelser under INPPL1- och PPP6R2-mRNA-splitsning. Dessa fynd indikerar den potentiella betydelsen av G4-strukturen i SF3B4-medierad alternativ splitsning.
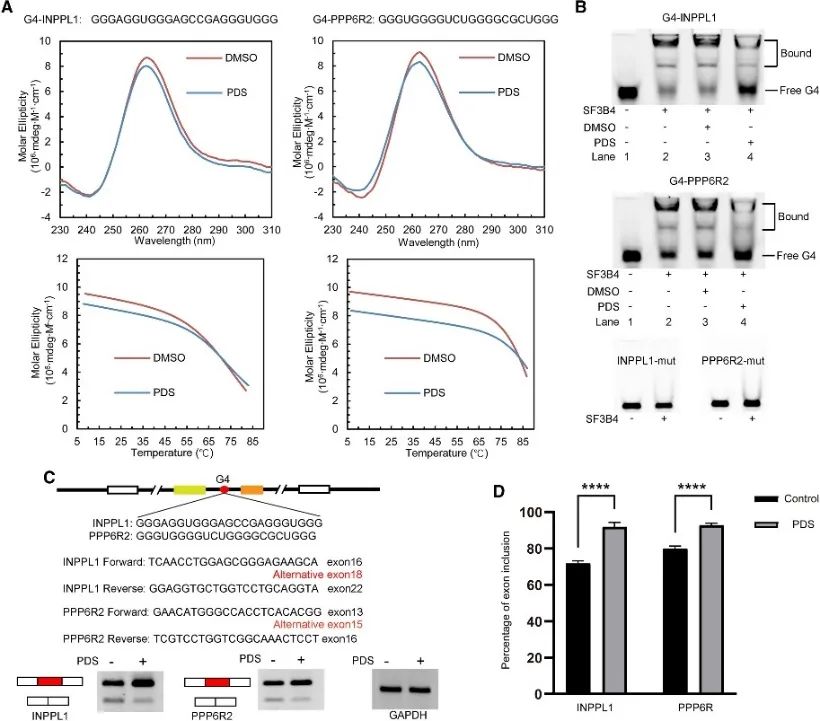
Figur 6 SF3B4 binder G4-strukturen och påverkar den alternativa splitsningen av mRNA.
Sammanfattningsvis introducerar denna studie en effektiv verktygslåda som omfattar G4PID-proben och PLGPB-strategin, som belyser hur samspelet mellan G4 och proteiner i cellen påverkar genuttrycket, vilket särskilt påverkar den centrala processen med alternativ splitsning. Dessa fynd fördjupar vår förståelse av de reglerande mekanismerna som involverar G4 i cellulära biologiska processer och banar väg för ytterligare utforskning av terapeutiska strategier för G4-relaterade sjukdomar.

